El archivo contiene un grupo de funciones usadas para procesar los datos de este prototipo, se separaron del bloque principal de instrucciones con el objetivo de simplificar el desarrollo del las actividades principales.
Recuerde disponer de este archivo en la misma carpeta que el algoritmo que invoca a la librería.
archivo de libreria: girni_lorawan_libreria
Instrucciones Python
# Girni LoRaWan librerias 2021-10-28
# LoRaWan, lecturas de Rssi y SNR
# Propuesta: edelros@espol.edu.ec
# http://blog.espol.edu.ec/girni/
import numpy as np
import json
import pandas as pd
import datetime as dt
import os
import matplotlib.pyplot as plt
import matplotlib.dates as mdates
import matplotlib.units as munits
import geopandas as gpd
import fiona
import utm
def tablapunto(unarchivoDB,unsensor,gatewayDB,fechainicio,fechafin,zonaGMT=0):
'''Lectura de archivo.json hacia una tabla0
selecciona registros de "un sensor"
graba unreporte.csv con pandas
'''
campos = ['domain','entity_id','state','attributes','created']
# Lectura de archivo json, cambia a formato DataFrame
tabla0 = pd.DataFrame()
archivoexiste = os.path.exists(unarchivoDB)
if archivoexiste:
tabla0 = pd.read_json(unarchivoDB)
tabla0 = pd.DataFrame(tabla0,columns=campos)
else:
print(' ERROR: NO se encuentra el archivo...')
print(' revise el nombre de archivo. ')
# Revisa cada registro
leidos = 0
tabla = {}
if archivoexiste:
# Intervalo de fecha como datetime
hora_desplaza = dt.timedelta(hours = abs(zonaGMT))
fechaformatoDB = '%Y-%m-%dT%H:%M:%S.%f'
fechaformato = '%Y-%m-%d %H:%M:%S.%f'
fechatxt = fechainicio[0:np.min([26,len(fechainicio)])]
fechainicio = dt.datetime.strptime(fechatxt,fechaformato)
fechatxt = fechafin[0:np.min([26,len(fechafin)])]
fechafin = dt.datetime.strptime(fechatxt,fechaformato)
# datos de trama
for cadaregistro in tabla0.index:
unatrama = tabla0['attributes'][cadaregistro]
trama_mqtt = json.loads(unatrama)
# selecciona registro por sensor, en fecha y con datos
cualsensor = (tabla0['entity_id'][cadaregistro] == unsensor)
unafecha = tabla0['created'][cadaregistro]
unafecha = unafecha[0:np.min([26,len(unafecha)])]
unafecha = dt.datetime.strptime(unafecha,fechaformato)
unafecha = unafecha - hora_desplaza
enfecha = (unafecha>=fechainicio) and (unafecha<=fechafin)
condatos = 'applicationID' in trama_mqtt.keys()
selecciona = cualsensor and condatos and enfecha
if (selecciona == True):
# datos en la mensaje MQTT
publishedAt = trama_mqtt["publishedAt"]
publishedAt = publishedAt[0:np.min([26,len(publishedAt)])]
publishedAt = dt.datetime.strptime(publishedAt,fechaformatoDB)
publishedAt = publishedAt - hora_desplaza
friendly_name = trama_mqtt["friendly_name"]
deviceName = trama_mqtt["deviceName"]
dispositivo = friendly_name.split('_')[2]
dr = trama_mqtt["dr"]
fCnt = trama_mqtt["fCnt"]
fPort = trama_mqtt["fPort"]
objectJSON = trama_mqtt["objectJSON"]
datosensor = json.loads(objectJSON)
freq_tx = trama_mqtt["txInfo"]["frequency"]
bandwidth = trama_mqtt["txInfo"]["loRaModulationInfo"]["bandwidth"]
spreadingFactor = trama_mqtt["txInfo"]["loRaModulationInfo"]["spreadingFactor"]
datostrama = {"publishedAt": publishedAt,
"dispositivo": dispositivo,
"fCnt": fCnt}
# revisa gateway en la red LoRaWan
tamano = len(trama_mqtt["rxInfo"])
i = 0
while i<tamano:
gatewayID = trama_mqtt["rxInfo"][i]["gatewayID"]
rssi = trama_mqtt["rxInfo"][i]["rssi"]
loRaSNR = trama_mqtt["rxInfo"][i]["loRaSNR"]
channel = trama_mqtt["rxInfo"][i]["channel"]
rfChain = trama_mqtt["rxInfo"][i]["rfChain"]
gtwNum = gatewayDB[gatewayID]
# registra en tabla, incluyendo tramas repetidas
datostrama['rssi_up'] = rssi
datostrama['snr_up'] = loRaSNR
datostrama['channel_up'] = channel
datostrama['rfChain_up'] = rfChain
datostrama['gtw_rx'] = gtwNum
i = i + 1
# dato del sensor
equivale = {'Down_datarate': 'dr_down',
'Down_rssi' : 'rssi_down',
'Down_snr' : 'snr_down',
'bateria_V' : 'bateria_V'}
for undato in datosensor:
if undato in equivale.keys():
datostrama[equivale[undato]] = datosensor[undato]
else:
datostrama[undato] = datosensor[undato]
# datos restantes
datostrama["frequency"] = freq_tx
datostrama["bandwidth"] = bandwidth
datostrama["spreadingFactor"] = spreadingFactor
datostrama["fPort"] = fPort
datostrama["dr"] = dr
datostrama["created"] = unafecha
leidos = leidos + 1
# revisa registro repetido
repetido = False
if leidos>1:
trama_num = (fCnt == tabla[leidos-1]['fCnt'])
fecha_pub = (publishedAt == tabla[leidos-1]['publishedAt'])
gtwNum_rep = (gtwNum == tabla[leidos-1]['gtw_rx'])
repetido = (trama_num and fecha_pub and gtwNum_rep)
if not(repetido):
tabla[leidos] = datostrama
else:
leidos = leidos - 1
# convierte diccionario en DataFrame
if len(tabla)>0:
tabla = pd.DataFrame(tabla)
tabla = tabla.T
return(tabla)
def describe_punto(tabla,codigopunto,carpeta_rsm,variables):
''' Descriptores estadisticos de los datos.csv
de un dispositivo, revisa media, desviació estándar, pmf
graba unreporte.csv con pandas y genera gráficas
'''
medida = variables['medida']
medida_unidad = variables['medida_unidad']
medida_normal = variables['medida_normal']
movAvg_cual = variables['movAvg_cual']
medida_grafica = variables['medida_grafica']
movAvg_color = variables['movAvg_color']
guarda = variables['guarda']
precision = variables['precision']
fechaformato = "%Y-%m-%d %H:%M:%S.%f"
# medida intervalo
medida_min = np.min(medida_normal)
medida_max = np.max(medida_normal)
# fechas series a datetime
tabla['publishedAt'] = pd.to_datetime(tabla['publishedAt'],
format=fechaformato)
tabla['created'] = pd.to_datetime(tabla['publishedAt'],
format=fechaformato)
# revisa errores de medida
tabla["error_up"] = 0
tabla["error_down"] = 0
for undato in tabla['publishedAt'].keys():
medida_up = tabla[medida+'_up'][undato]
enrango = (medida_up>=np.min(medida_normal))
enrango = (enrango and medida_up<=np.max(medida_normal))
if not(enrango):
tabla.at[undato,"error_up"] = 1
medida_down = tabla[medida+'_down'][undato]
enrango = (medida_down>=np.min(medida_normal))
enrango = (enrango and medida_down<=np.max(medida_normal))
if not(enrango):
tabla.at[undato,"error_down"] = 1
# tasa error trama
leidos = len(tabla)
if leidos > 0:
error_up = np.sum(tabla['error_up'])
error_up = error_up/leidos
error_down = np.sum(tabla['error_down'])
error_down = error_down/leidos
# descriptor estadístico, datos sin errores
condicion_up = (tabla['error_up']==0)
condicion_down = (tabla['error_down']==0)
medida_up = tabla[condicion_up][medida+'_up']
describe_up = medida_up.describe()
describe_up['error_trama'] = error_up
medida_down = tabla[condicion_down][medida+'_down']
describe_down = medida_down.describe()
describe_down['error_trama'] = error_down
descriptor = describe_up.copy()
descriptor = pd.concat([descriptor,describe_down],axis=1)
descriptor['dispositivo'] = tabla['dispositivo'][0]
pmf_up = medida_pmf(medida_up,describe_up)
pmf_down = medida_pmf(medida_down,describe_down)
pmf_punto = {'pmf':{'pmf_up' : pmf_up,
'pmf_down' : pmf_down}}
pmf_punto = pd.DataFrame(pmf_punto)
pmf_punto['dispositivo'] = tabla['dispositivo'][0]
# Para gráficas
# medias moviles en movAvg_cual[]
serie_up = pd.Series(medida_up)
movAvg_up_mean = []
movAvg_up_std = []
m = len(movAvg_cual)
for j in range(0,m,1):
k = movAvg_cual[j]
movAvg_up_mean.append(list(serie_up.rolling(k).mean()))
movAvg_up_std.append(list(serie_up.rolling(k).std()))
serie_down = pd.Series(medida_down)
movAvg_down_mean = []
movAvg_down_std = []
for j in range(0,m,1):
k = movAvg_cual[j]
movAvg_down_mean.append(list(serie_down.rolling(k).mean()))
movAvg_down_std.append(list(serie_down.rolling(k).std()))
movAvgData ={'movAvg_cual' : movAvg_cual,
'movAvg_color' : movAvg_color,
'movAvg_up_mean' : movAvg_up_mean,
'movAvg_down_mean': movAvg_down_mean,
'movAvg_up_std' : movAvg_up_std,
'movAvg_down_std' : movAvg_down_std
}
grafData ={'codigopunto' : codigopunto,
'medida' : medida,
'precision': precision,
'medida_unidad' : medida_unidad,
'medida_grafica': medida_grafica
}
resultado = [tabla,descriptor,pmf_punto,movAvgData,grafData]
return(resultado)
# función de probabilidad de masa pmf
def medida_pmf(valores,undescriptor):
pmin = np.min(valores)
pmax = np.max(valores)
tramo = int(pmax-pmin)
conteo = np.zeros(tramo+1,dtype=int)
intervalo = np.arange(pmin,pmax+1,1)
for valor in valores:
donde = np.where(intervalo == valor)
conteo[donde] = conteo[donde] + 1
freq_relativa = np.array(conteo)/np.sum(conteo)
unpmf = {'intervalo' : list(intervalo),
'freq_relativa' : list(freq_relativa)}
return(unpmf)
# GRAFICA -----
def graf_puntos_serie(tabla,descriptor,movAvgData,grafData):
''' grafica la serie de tiempo de cada punto
añade medias móviles
'''
# ajuste de formato de fecha para eje x
converter = mdates.ConciseDateConverter()
munits.registry[np.datetime64] = converter
munits.registry[dt.date] = converter
munits.registry[dt.datetime] = converter
# datos para grafica
precision = grafData['precision']
medida = grafData['medida']
medida_unidad = grafData['medida_unidad']
medida_grafica = grafData['medida_grafica']
movAvg_cual = movAvgData['movAvg_cual']
movAvg_color = movAvgData['movAvg_color']
media_up = descriptor[medida+'_up']['mean']
std_up = descriptor[medida+'_up']['std']
media_down = descriptor[medida+'_down']['mean']
std_down = descriptor[medida+'_down']['std']
# ajuste de intervalo eje y
y_min = np.min([np.min(medida_grafica),
media_up - 2*std_up,
media_down - 2*std_down])
y_max = np.max([np.max(medida_grafica),
media_up + 2*std_up,
media_down + 2*std_down])
# selecciona sin error
condicion_up = (tabla['error_up']==0)
condicion_down = (tabla['error_down']==0)
# grafica
fig_serie,(graf_up,graf_down) = plt.subplots(2,1)
# medida_up -----
graf_up.plot(tabla[condicion_up]['publishedAt'],
tabla[condicion_up][medida+'_up'],
color='blue',marker ='.',
linestyle='')
# medida_up, medias y std
etiq_up = str(np.round(media_up,precision))+' +/- '
etiq_up = etiq_up + str(np.round(std_up,precision))
graf_up.axhline(media_up,
color='blue',label=etiq_up)
graf_up.axhline(media_up-std_up,
color='blue',linestyle='dotted')
graf_up.axhline(media_up+std_up,
color='blue',linestyle='dotted')
# medida_up, medias móviles
m = len(movAvg_cual)
for j in range(0,m,1):
k = str(movAvg_cual[j])
graf_up.plot(tabla[condicion_up]['publishedAt'],
movAvgData['movAvg_up_mean'][j],
label='movAvg_'+k,
color=movAvg_color[j])
graf_up.set_ylim(y_min,y_max)
graf_up.set_ylabel(medida+'_up ('+medida_unidad+')',
color='blue')
graf_up.legend()
graf_up.grid(True,linestyle='dotted',
axis='x',which='both')
# medida_down -------
graf_down.plot(tabla[condicion_down]['publishedAt'],
tabla[condicion_down][medida+'_down'],
color='brown',marker ='.',
linestyle='')
# medida_down, medias y std
etiq_down = str(np.round(media_down,precision))+' +/- '
etiq_down = etiq_down + str(np.round(std_down,precision))
graf_down.axhline(media_down,
color='brown',label=etiq_down)
graf_down.axhline(media_down+std_down,
color='brown',linestyle='dotted')
graf_down.axhline(media_down-std_down,
color='brown',linestyle='dotted')
# medida_down, medias moviles
for j in range(0,m,1):
k = str(movAvg_cual[j])
graf_down.plot(tabla[condicion_down]['publishedAt'],
movAvgData['movAvg_down_mean'][j],
label='movAvg_'+k,
color=movAvg_color[j])
graf_down.set_ylim(y_min,y_max)
graf_down.set_xlabel('fecha')
graf_down.set_ylabel(medida+'_down ('+medida_unidad+')',
color='brown')
graf_down.legend()
graf_down.grid(True,linestyle='dotted',
axis='x', which='both')
graf_up.set_title('Serie: '+grafData['codigopunto']+' '+ medida)
plt.tight_layout()
return(fig_serie)
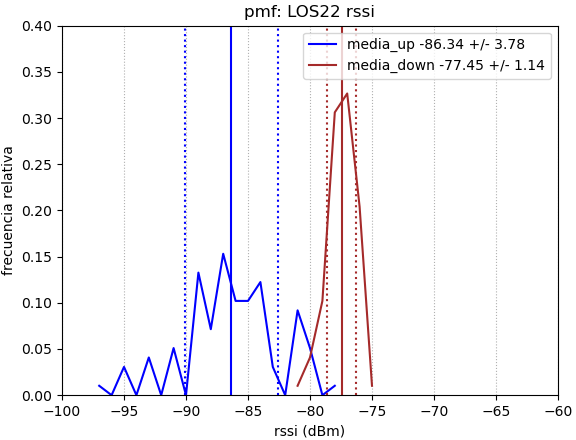
def graf_puntos_pmf(pmf_punto,descriptor,grafData):
''' grafica función de probabilida de masa
para cada punto, media +/- std
'''
# datos para grafica
x_pmfup = pmf_punto['pmf']['pmf_up']['intervalo']
y_pmfup = pmf_punto['pmf']['pmf_up']['freq_relativa']
x_pmfdown = pmf_punto['pmf']['pmf_down']['intervalo']
y_pmfdown = pmf_punto['pmf']['pmf_down']['freq_relativa']
precision = grafData['precision']
medida = grafData['medida']
medida_unidad = grafData['medida_unidad']
medida_grafica = grafData['medida_grafica']
media_up = descriptor[medida+'_up']['mean']
std_up = descriptor[medida+'_up']['std']
media_down = descriptor[medida+'_down']['mean']
std_down = descriptor[medida+'_down']['std']
prob_max = 0.40
# ajuste de intervalo eje y
y_min = np.min([np.min(medida_grafica),
media_up - 2*std_up,
media_down - 2*std_down])
y_max = np.max([np.max(medida_grafica),
media_up + 2*std_up,
media_down + 2*std_down])
# grafica
fig_pmf,graf_pmf = plt.subplots()
etiq_up = str(np.round(media_up,precision)) +' +/- '
etiq_up = etiq_up + str(np.round(std_up,precision))
graf_pmf.plot(x_pmfup,y_pmfup,
label='media_up '+etiq_up,
color='blue')
graf_pmf.axvline(media_up,color='blue')
graf_pmf.axvline(media_up+std_up,
linestyle='dotted',color='blue')
graf_pmf.axvline(media_up-std_up,
linestyle='dotted',color='blue')
etiq_down = str(np.round(media_down,precision))+' +/- '
etiq_down = etiq_down + str(np.round(std_down,precision))
graf_pmf.plot(x_pmfdown,y_pmfdown,
label='media_down '+etiq_down,
color='brown')
graf_pmf.axvline(media_down,color='brown')
graf_pmf.axvline(media_down+std_down,
linestyle='dotted',color='brown')
graf_pmf.axvline(media_down-std_down,
linestyle='dotted',color='brown')
graf_pmf.set_title('pmf: '+grafData['codigopunto']+' '+medida)
graf_pmf.set_xlim(y_min,y_max)
graf_pmf.set_ylim(0,prob_max)
graf_pmf.set_xlabel(medida+' ('+medida_unidad+')')
graf_pmf.set_ylabel('frecuencia relativa')
graf_pmf.legend()
graf_pmf.grid(True,linestyle='dotted',
axis='x', which='both')
return(fig_pmf)
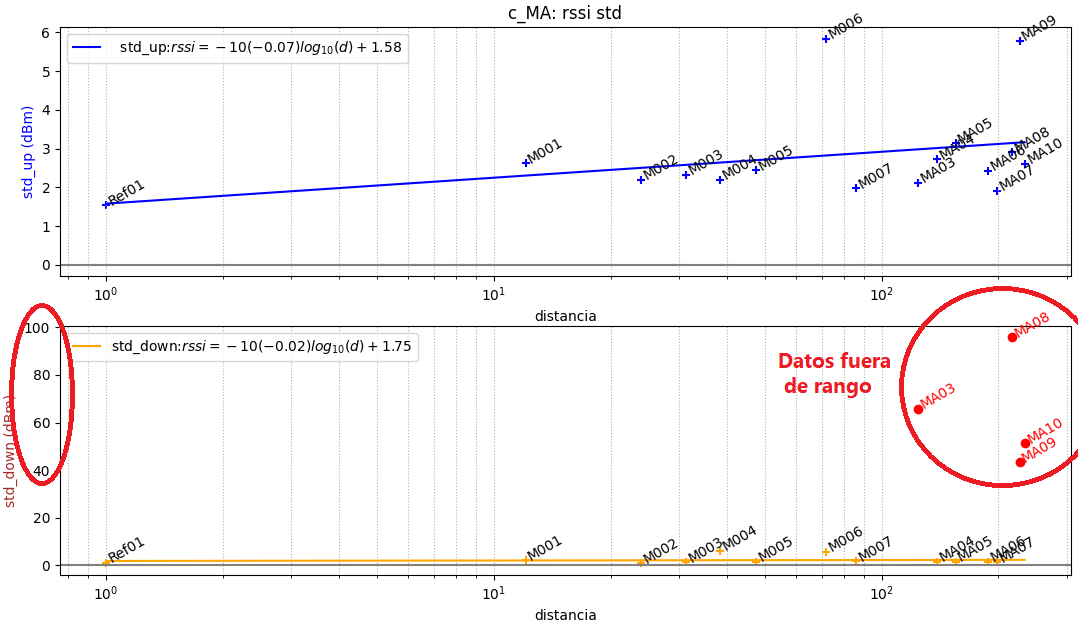
def graf_puntos_std(tabla,descriptor,movAvgData,grafData):
''' grafica serie de std usando medias moviles
para cada punto, media_std
'''
# ajuste de formato de fecha para eje x
converter = mdates.ConciseDateConverter()
munits.registry[np.datetime64] = converter
munits.registry[dt.date] = converter
munits.registry[dt.datetime] = converter
# datos para grafica
precision = grafData['precision']
medida = grafData['medida']
medida_unidad = grafData['medida_unidad']
movAvg_cual = movAvgData['movAvg_cual']
movAvg_color = movAvgData['movAvg_color']
# selecciona sin error
condicion_up = (tabla['error_up']==0)
condicion_down = (tabla['error_down']==0)
# ajuste de intervalo eje y
y_min = 0
y_max = np.max([2, 2*descriptor[medida+'_up']['std'],
2*descriptor[medida+'_down']['std']])
# grafica
fig_std,(graf_stdUp,graf_stdDown) = plt.subplots(2,1)
# std up
std_up = np.round(descriptor[medida+'_up']['std'],precision)
graf_stdUp.axhline(std_up,label='std '+str(std_up),
color='blue')
m = len(movAvg_cual)
for j in range(0,m,1):
k = str(movAvg_cual[j])
graf_stdUp.plot(tabla[condicion_up]['publishedAt'],
movAvgData['movAvg_up_std'][j],
label='movAvg_'+k,
color=movAvg_color[j])
graf_stdUp.set_ylim(y_min,y_max)
graf_stdUp.set_ylabel('std_up ('+medida_unidad+')',
color='blue')
graf_stdUp.legend()
graf_stdUp.grid(True,linestyle='dotted',
axis='x', which='both')
graf_stdUp.set_title('std: '+grafData['codigopunto']+' '+ medida)
# std down
std_down = np.round(descriptor[medida+'_down']['std'],precision)
graf_stdDown.axhline(std_down,label='std '+str(std_down),
color='brown')
for j in range(0,m,1):
k = str(movAvg_cual[j])
graf_stdDown.plot(tabla[condicion_down]['publishedAt'],
movAvgData['movAvg_down_std'][j],
label='movAvg_'+k,color=movAvg_color[j])
graf_stdDown.set_ylim(y_min,y_max)
graf_stdDown.set_xlabel('fecha')
graf_stdDown.set_ylabel('std_down ('+medida_unidad+')',
color='brown')
graf_stdDown.legend()
graf_stdDown.grid(True,linestyle='dotted',
axis='x', which='both')
plt.tight_layout()
return(fig_std)
def linealiza_lstsq(xi,yi,digitos = 3):
''' usa minimos cuadrados para entregar la ecuacion
digitos: usados en expresion latex
'''
unaecuacion = {}
# Eje x en log10()
xilog = np.log10(xi)
n = len(xi)
# mínimos cuadrados (least square),
# distancia vs medida
A = np.vstack([xilog, np.ones(n)]).T
[m0, b0] = np.linalg.lstsq(A, yi, rcond=None)[0]
alpha = -m0/10
beta = b0
# coeficiente de correlación
coeficientes = np.corrcoef(xilog,yi)
r = coeficientes[0,1]
# coeficiente de determinación
r2 = r**2
# ecuaciones expresion rssi(d)
fdist0 = lambda d: -10*alpha*(np.log10(d))+beta
fdtxt0 = r'$ rssi = -10(' + str(np.round(alpha,digitos))
fdtxt0 = fdtxt0 + ')log_{10}(d)' # +('
texto = '+'
if beta <0:
texto = '-'
fdtxt0 = fdtxt0 + texto + str(np.round(np.abs(beta),digitos))+' $'
# Errores respecto a rssi(d)
yi0 = fdist0(xi)
dyi0 = yi - yi0
dyi0mean = np.mean(np.abs(dyi0))
dyi0std = np.std(dyi0, dtype=np.float64)
# ecuaciones expresion d(rssi)
grssi0 = lambda rssi: 10**((beta-rssi)/(10*alpha))
grtxt0 = r"$ d = 10^{(" + str(np.round(beta,digitos)) + ' - '
grtxt0 = grtxt0 + 'rssi)/' + '(10('+str(np.round(alpha,digitos))+'))} $'
# Errores respecto a rssi(d)
xi0 = grssi0(yi)
dxi0 = xi - xi0
dxi0mean = np.mean(np.abs(dxi0))
dxi0std = np.std(dxi0, dtype=np.float64)
yimin = np.around(np.min(yi),2)
yimax = np.around(np.max(yi),2)
unaecuacion = {'alpha' : alpha,
'beta' : beta,
'coef_correlacion' : r,
'coef_determinacion' : r2,
'eq_latex': fdtxt0,
'intervalox' : [np.min(xi),np.max(xi)],
'error_medio': dyi0mean,
'error_std' : dyi0std,
'eqg_latex' : grtxt0,
'intervaloy' : [yimin,yimax],
'errorx_medio': dxi0mean,
'errorx_std' : dxi0std,
}
return(unaecuacion)
def ecuacion_gradiente(carpeta_rsm,arch_coord,var_gen):
''' desarrolla la ecuación para un gradiente
'''
var_gen['movAvg_cual'] = [2,4] #cada cuantas muestras
partes = carpeta_rsm.strip('/').split('_')
arch_nombre = partes[1]+'_'+partes[2]
carp_coord = var_gen['carp_coord']
medida = var_gen['medida']
precision = var_gen['precision']
# lee coordenadas y su distancia al gateway
dist_Gtw = coordenadas_leer(arch_coord,carp_coord)
# crea lista.txt de archivos a usar si no existe
arch_lista = medida+"_"+arch_nombre+"_lista.txt"
archivo_ruta = carpeta_rsm + '/' + arch_lista
archivoexiste = os.path.exists(archivo_ruta)
if not(archivoexiste):
puntoUsar = pd.DataFrame(columns=['punto', 'up', 'down'])
for unarchivo in os.listdir(carpeta_rsm):
verifica = unarchivo.startswith('describe_')
verifica = verifica and unarchivo.endswith('.csv')
if verifica:
partes = unarchivo.strip('.csv').split('_')
unpunto = partes[2]
puntoUsar = puntoUsar.append({'punto' : unpunto,
'up' : 1,
'down' : 1},
ignore_index = True)
puntoUsar = puntoUsar.set_index('punto')
puntoUsar.to_csv(archivo_ruta)
# usa lista.txt de archivos seleccionados 1 ó 0 para enlace up,down
if (archivoexiste):
puntoUsar = pd.read_csv(archivo_ruta)
puntoUsar = puntoUsar.set_index('punto')
# Datos para gráfica desde lista.txt
punto_graf = pd.DataFrame()
puntoSinDist = []
for unarchivo in os.listdir(carpeta_rsm):
if unarchivo.startswith('describe_'):
codigopunto = unarchivo.strip('.csv').split('_')[2]
# lectura del archivo
unresumen = carpeta_rsm+'/'+unarchivo
descriptor = pd.read_csv(unresumen,index_col='Unnamed: 0')
if (codigopunto in dist_Gtw.index):
undato = {'codigopunto': codigopunto,
'dist_Gtw' : dist_Gtw[codigopunto],
medida+'_up' : descriptor['rssi_up']['mean'],
medida+'_up'+'_std': descriptor['rssi_up']['std'],
'usar_up' : puntoUsar['up'][codigopunto],
medida+'_down': descriptor['rssi_down']['mean'],
medida+'_down'+'_std' :descriptor['rssi_down']['std'],
'usar_down' : puntoUsar['down'][codigopunto],
'dispositivo': descriptor['dispositivo'][0]
}
punto_graf = punto_graf.append(undato,ignore_index=True)
else:
puntoSinDist.append(codigopunto)
punto_graf = punto_graf.set_index('codigopunto')
punto_graf = punto_graf.sort_values('dist_Gtw' )
# selecciona datos
dist = punto_graf['dist_Gtw']
usar_up = punto_graf['usar_up']
usar_down = punto_graf['usar_down']
# Eje x en log10()
xi = np.array(dist)
# enlace_up
media_up = punto_graf[medida+'_up']
std_up = punto_graf[medida+'_up'+'_std']
media_up_techo = media_up + std_up
media_up_piso = media_up - std_up
# ecuacion enlace up
yi_up = np.array(media_up)
eq_up = linealiza_lstsq(xi[usar_up==1],
yi_up[usar_up==1],
digitos = precision)
alpha_up = eq_up['alpha']
beta_up = eq_up['beta']
fd_up = lambda d: -10*alpha_up*(np.log10(d))+beta_up
# errores up de puntos vs ecuacion
fi_up = fd_up(xi)
dyi0std = eq_up['error_std']
punto_graf['fi_up'] = fi_up
# enlace_down
media_down = punto_graf[medida+'_down']
std_down = punto_graf[medida+'_down'+'_std']
media_down_techo = media_down + std_down
media_down_piso = media_down - std_down
# ecuación Enlace down
yi_down = np.array(media_down)
eq_down = linealiza_lstsq(xi[usar_down==1],
yi_down[usar_down==1],
digitos = precision)
alpha_down = eq_down['alpha']
beta_down = eq_down['beta']
fd_down = lambda d: -10*alpha_down*(np.log10(d))+beta_down
# errores down de puntos vs ecuacion
fi_down = fd_down(xi)
dyi1std = eq_down['error_std']
punto_graf['fi_down'] = fi_down
resultado = {'tabla' : punto_graf,
'ecuacion_up' : eq_up,
'ecuacion_down' : eq_down,
'puntoSinDist' : puntoSinDist}
return (resultado)
def coordenadas_leer(arch_coord,carp_coord):
''' lista las coordenadas desde un archivo
ubicado en la carpeta
'''
if len(carp_coord)>0:
carp_coord = carp_coord+'/'
archivo_ruta = carp_coord+arch_coord
arch_coord_existe = os.path.exists(archivo_ruta)
if arch_coord_existe:
puntos = pd.read_csv(archivo_ruta,index_col='punto')
# busca gateway y ubicación de punto observado
Gw_nombre = ''
for unpunto in puntos.index:
if unpunto.startswith('Gw'):
Gw_nombre = unpunto
dist_Gtw = puntos['dist_'+Gw_nombre]
else:
print(' ERROR: NO se encuentra el archivo...')
print(' revise el ruta/nombre de archivo. ')
return(dist_Gtw)
def graf_gradiente(arch_nombre,punto_graf,ecuacion,var_gen):
''' grafica de un gradiente
'''
escalabase = 10 # 10
escala = 'log'
medida = var_gen['medida']
precision = var_gen['precision']
# enlace_up
media_up = punto_graf[medida+'_up']
std_up = punto_graf[medida+'_up'+'_std']
media_up_techo = media_up + std_up
media_up_piso = media_up - std_up
yi_up = np.array(media_up)
fi_up = punto_graf['fi_up']
# enlace_down
media_down = punto_graf[medida+'_down']
std_down = punto_graf[medida+'_down'+'_std']
media_down_techo = media_down + std_down
media_down_piso = media_down - std_down
yi_down = np.array(media_down)
fi_down = punto_graf['fi_down']
dist = punto_graf['dist_Gtw']
usar_up = punto_graf['usar_up']
usar_down = punto_graf['usar_down']
eq_up = ecuacion['ecuacion_up']
eq_down = ecuacion['ecuacion_down']
dyi0std = eq_up['error_std']
dyi1std = eq_down['error_std']
fig_gradnt,(graf_up,graf_down) = plt.subplots(2,1)
if escala == 'log':
graf_up.set_xscale(escala,base=escalabase)
graf_down.set_xscale(escala,base=escalabase)
# medida up +/- std
graf_up.scatter(dist,media_up,color='blue',marker='o')
graf_up.scatter(dist,media_up_techo,color='blue',marker='+')
graf_up.scatter(dist,media_up_piso,color='blue',marker='+')
# medida down +/- std
graf_down.scatter(dist,media_down,color='orange',marker='o')
graf_down.scatter(dist,media_down_techo,color='orange',marker='+')
graf_down.scatter(dist,media_down_piso,color='orange',marker='+')
# linealizado up
graf_up.plot(dist,fi_up,label = ' up:'+eq_up['eq_latex'],
color='blue')
graf_up.plot(dist,fi_up+dyi0std,
color='blue',linestyle='dotted')
graf_up.plot(dist,fi_up-dyi0std,
color='blue',linestyle='dotted')
# linealizado down
graf_down.plot(dist,fi_down,label = 'down:'+eq_down['eq_latex'],
color='orange')
graf_down.plot(dist,fi_down+dyi1std,
color='orange',linestyle='dotted')
graf_down.plot(dist,fi_down-dyi1std,
color='orange',linestyle='dotted')
# ajuste de eje y
y_min = np.min([np.min(media_up_piso),np.min(media_down_piso)])
y_max = np.max([np.max(yi_up+dyi0std),np.max(yi_down+dyi1std)])
if y_min<-135:
y_min = np.min([np.min([media_up]),np.min([media_down])])
graf_up.set_ylim(y_min,y_max)
graf_down.set_ylim(y_min,y_max)
# etiquetas up
for unpunto in punto_graf.index:
media_etiq = np.round(punto_graf[medida+'_up'][unpunto],precision)
usarpunto = punto_graf['usar_up'][unpunto]
etiqueta = unpunto #+str(media_etiq)
xi = punto_graf['dist_Gtw'][unpunto]
yi = punto_graf['rssi_up'][unpunto]
if usarpunto:
graf_up.annotate(etiqueta,(xi,yi), rotation=45)
if not(usarpunto):
graf_up.annotate(etiqueta,(xi,yi), rotation=45,color='red')
graf_up.scatter(dist[unpunto],media_up[unpunto],color='red',marker='o')
graf_up.scatter(dist[unpunto],media_up_techo[unpunto],
color='red',marker='+')
# etiquetas down
for unpunto in punto_graf.index:
media_etiq = np.round(punto_graf[medida+'_down'][unpunto],precision)
usarpunto = punto_graf['usar_down'][unpunto]
etiqueta = unpunto #+str(media_etiq)
xi = punto_graf['dist_Gtw'][unpunto]
yi = punto_graf['rssi_down'][unpunto]
if usarpunto:
graf_down.annotate(etiqueta,(xi,yi), rotation=45)
if not(usarpunto):
graf_down.annotate(etiqueta,(xi,yi), rotation=45,color='red')
graf_down.scatter(dist[unpunto],media_down[unpunto],color='red',marker='o')
graf_down.scatter(dist[unpunto],media_down_techo[unpunto],
color='red',marker='+')
graf_up.set_ylabel(medida+'_up (dBm)',color='blue')
graf_up.set_title(arch_nombre+' '+medida)
graf_up.legend()
graf_up.grid(True,linestyle='dotted',
axis='x', which='both')
graf_down.set_xlabel('distancia')
graf_down.set_ylabel(medida+'_down (dBm)',color='brown')
graf_down.legend()
graf_down.grid(True,linestyle='dotted',
axis='x', which='both')
return(fig_gradnt)
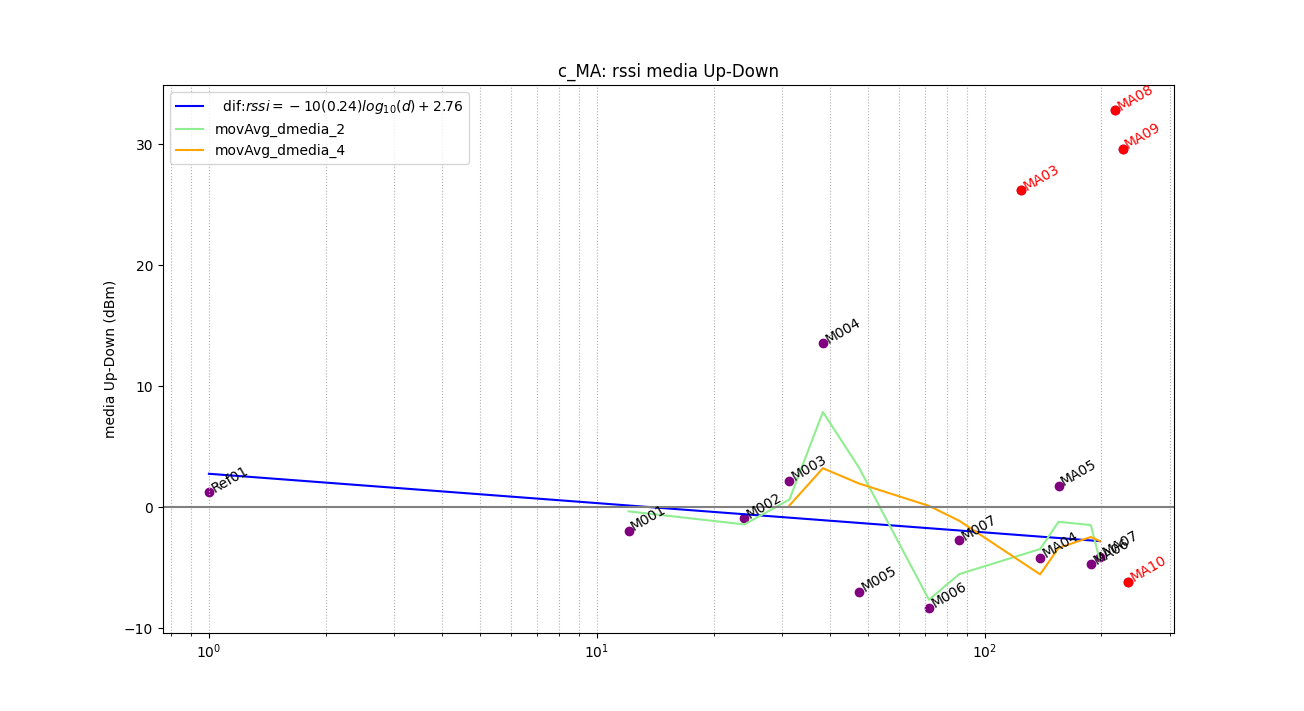
def graf_diferencia_updown(arch_nombre,punto_graf,var_gen):
'''Grafica de diferencias up-down
'''
medida = var_gen['medida']
medida_unidad = var_gen['medida_unidad']
movAvg_cual = var_gen['movAvg_cual']
movAvg_color = var_gen['movAvg_color']
precision = var_gen['precision']
# analiza diferencias de medias y std --------------
punto_graf['usar_dif'] = punto_graf['usar_up']*punto_graf['usar_down']
usar_dif = punto_graf['usar_dif']
punto_graf['dif_mean'] = punto_graf['rssi_up']-punto_graf['rssi_down']
punto_graf['dif_std'] = punto_graf[medida+'_up_std']-punto_graf[medida+'_down_std']
punto_graf['dif_top'] = punto_graf['dif_mean']+punto_graf['dif_std']
punto_graf['dif_bottom'] = punto_graf['dif_mean']-punto_graf['dif_std']
dist = punto_graf['dist_Gtw']
usar_dif = punto_graf['usar_dif']
# linealiza diferencia
xi_dif = np.array(punto_graf['dist_Gtw'][usar_dif==1])
yi_dif = np.array(punto_graf['dif_mean'][usar_dif==1])
eq_dif = linealiza_lstsq(xi_dif,yi_dif,digitos = precision)
alpha_dif = eq_dif['alpha']
beta_dif = eq_dif['beta']
fdistdif0 = lambda d: -10*alpha_dif*(np.log10(d))+beta_dif
yi_dif0 = fdistdif0(xi_dif)
ecua_dif = pd.DataFrame(eq_dif)
# medias moviles en movAvg_cual[]
serie_media = pd.Series(punto_graf['dif_mean'][usar_dif==1])
movAvg_dmedia = []
m = len(movAvg_cual)
for j in range(0,m,1):
k = movAvg_cual[j]
movAvg_dmedia.append(serie_media.rolling(k).mean())
# Grafica diferencia
escalabase = 10 # 10
escala = 'log'
fig_dif_updown,graf_dmean = plt.subplots()
if escala == 'log':
graf_dmean.set_xscale(escala,base=escalabase)
# diferencia medida up - down
graf_dmean.scatter(dist,punto_graf['dif_mean'],color='purple',marker='o')
graf_dmean.plot(xi_dif,yi_dif0,label = ' dif:'+eq_dif['eq_latex'],color='blue')
graf_dmean.set_ylabel('media Up-Down (dBm)')
# medida_up, medias móviles
for j in range(0,m,1):
k = str(movAvg_cual[j])
graf_dmean.plot(dist[usar_dif==1],movAvg_dmedia[j],
label='movAvg_dmedia_'+k,color=movAvg_color[j])
# etiquetas
i=0
for unpunto in punto_graf.index:
media_etiq = np.round(punto_graf[medida+'_up'][unpunto],precision)
etiqueta = unpunto # + ' ' +str(media_etiq)
usar_dif = punto_graf['usar_dif'][unpunto]
xi = punto_graf['dist_Gtw'][unpunto]
yi = punto_graf.iloc[i]['dif_mean']
i = i+1
if usar_dif:
graf_dmean.annotate(etiqueta,(xi,yi), rotation=30)
if not(usar_dif):
graf_dmean.annotate(etiqueta,(xi,yi),color='red',rotation=30)
graf_dmean.scatter(xi,yi,color='red',marker='o')
graf_dmean.axhline(0,color='grey')
graf_dmean.grid(True,linestyle='dotted',axis='x', which='both')
graf_dmean.legend()
graf_dmean.set_title(arch_nombre+': '+medida+' media Up-Down')
return(fig_dif_updown)
def graf_gradiente_std(arch_nombre,punto_graf,medida,precision):
''' Grafica de std_up y std_down ------
'''
xi_std = np.array(punto_graf['dist_Gtw'])
yi_std_up = np.array(punto_graf[medida+'_up_std'])
yi_std_down = np.array(punto_graf[medida+'_down_std'])
# linealiza std
usar_up = punto_graf['usar_up']
usar_down = punto_graf['usar_down']
eq_std_up = linealiza_lstsq(xi_std[usar_up==1],
yi_std_up[usar_up==1],
digitos = precision)
alpha_std_up = eq_std_up['alpha']
beta_std_up = eq_std_up['beta']
fdiststd_up0 = lambda d: -10*alpha_std_up*(np.log10(d))+beta_std_up
yi_std_up0 = fdiststd_up0(xi_std)
eq_std_down = linealiza_lstsq(xi_std[usar_down==1],
yi_std_down[usar_down==1],
digitos = precision)
alpha_std_down = eq_std_down['alpha']
beta_std_down = eq_std_down['beta']
fdiststd_down0 = lambda d: -10*alpha_std_down*(np.log10(d))+beta_std_down
yi_std_down0 = fdiststd_down0(xi_std)
# grafica std
escalabase = 10 # 10
escala = 'log'
fig_grad_std,(graf_std_up,graf_std_down) = plt.subplots(2,1)
if escala == 'log':
graf_std_up.set_xscale(escala,base=escalabase)
graf_std_down.set_xscale(escala,base=escalabase)
graf_std_up.plot(xi_std,yi_std_up0,
label = ' std_up:'+eq_std_up['eq_latex'],
color='blue')
# medida up +/- std
graf_std_up.scatter(xi_std,yi_std_up,color='blue',marker='+')
graf_std_down.scatter(xi_std,yi_std_down,color='orange',marker='+')
# etiquetas
i=0
for unpunto in punto_graf.index:
media_etiq = np.round(punto_graf[medida+'_up'][unpunto],precision)
etiqueta = unpunto # + ' ' +str(media_etiq)
usar_up = punto_graf['usar_up'][unpunto]
usar_down = punto_graf['usar_down'][unpunto]
xi = punto_graf['dist_Gtw'][unpunto]
yiu = punto_graf.iloc[i][medida+'_up_std']
yid = punto_graf.iloc[i][medida+'_down_std']
i = i+1
if usar_up:
graf_std_up.annotate(etiqueta,(xi,yiu), rotation=30)
if not(usar_up):
graf_std_up.annotate(etiqueta,(xi,yiu),color='red',rotation=30)
graf_std_up.scatter(xi,yiu,color='red',marker='o')
if usar_down:
graf_std_down.annotate(etiqueta,(xi,yid), rotation=30)
if not(usar_down):
graf_std_down.annotate(etiqueta,(xi,yid),color='red',rotation=30)
graf_std_down.scatter(xi,yid,color='red',marker='o')
graf_std_down.plot(xi_std,yi_std_down0,
label = 'std_down:'+eq_std_down['eq_latex'],
color='orange')
# otras etiquetas
graf_std_up.set_ylabel('std_up (dBm)', color='blue')
graf_std_up.set_xlabel('distancia')
graf_std_up.axhline(0,color='grey')
graf_std_up.grid(True,linestyle='dotted',
axis='x', which='both')
graf_std_up.set_title(arch_nombre+': '+medida+' std')
graf_std_up.legend()
graf_std_down.set_ylabel('std_down (dBm)', color='brown')
graf_std_down.set_xlabel('distancia')
graf_std_down.axhline(0,color='grey')
graf_std_down.grid(True,linestyle='dotted',
axis='x', which='both')
graf_std_down.legend()
return(fig_grad_std)
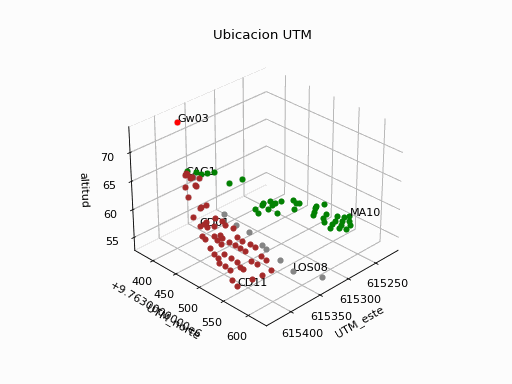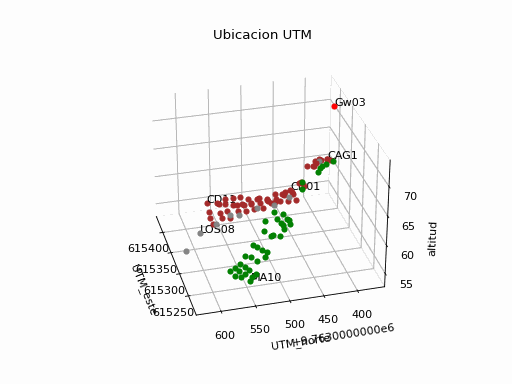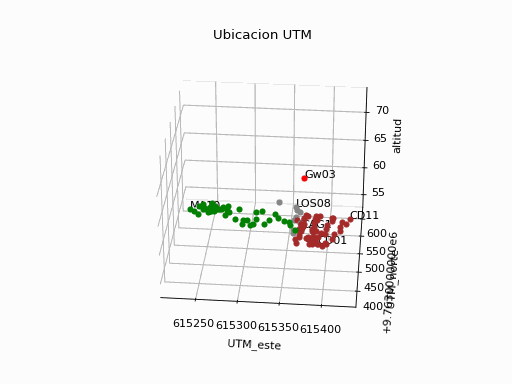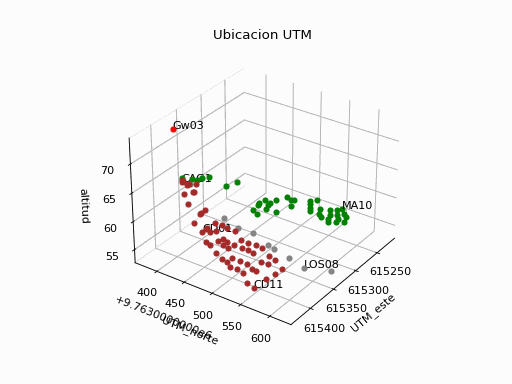
def coordenadas_kml_utm(archivo_ruta,zonaNum,zonaLetra,precision):
# Lectura de archivo
gpd.io.file.fiona.drvsupport.supported_drivers['KML'] = 'rw'
puntos = gpd.read_file(archivo_ruta, driver='KML')
puntos['dist'] = 0
tabla = pd.DataFrame()
for i in puntos.index:
# en latitud, longitud y altura
punto_nombre = puntos.loc[i]['Name']
longitud = puntos.loc[i]['geometry'].x
latitud = puntos.loc[i]['geometry'].y
altitud = puntos.loc[i]['geometry'].z
altitud = np.round(altitud,precision)
#en UTM
coord_utm = utm.from_latlon(latitud,longitud,
zonaNum,zonaLetra)
utm_este = np.round(coord_utm[0],precision)
utm_norte = np.round(coord_utm[1],precision)
# distancias a Gateways
dist = 0.0
if punto_nombre.startswith('Gw'):
n_Gtw = punto_nombre
x1 = utm_este
y1 = utm_norte
else:
x2 = utm_este
y2 = utm_norte
dist = np.sqrt((x2-x1)**2 + (y2-y1)**2)
dist = np.round(dist,precision)
unpunto = {'punto' : punto_nombre,
'utm_este' : utm_este,
'utm_norte' : utm_norte,
'utm_zNum' : int(zonaNum),
'utm_zLetra' : zonaLetra,
'altitud' : altitud,
'altitud_gps' : altitud,
'dist_'+n_Gtw : dist,
'latitud' : latitud,
'longitud' : longitud
}
tabla = tabla.append(unpunto,ignore_index=True)
tabla = tabla.set_index('punto')
return(tabla)
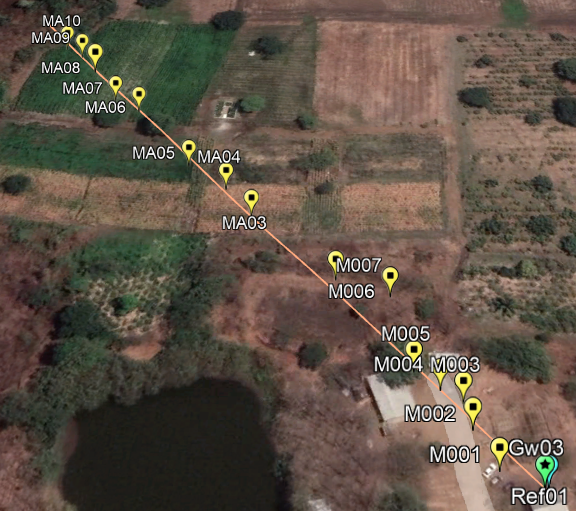
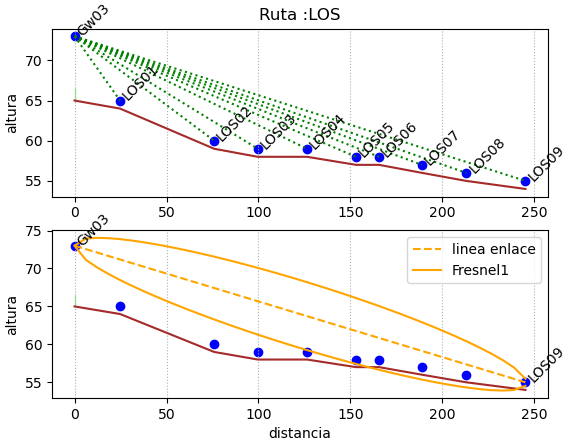
def revisa_perfil(archivo_ruta,alturaGw,alturapunto,
plantas,muestras=41,guarda=False):
LineaObs = archivo_ruta.strip('.csv').split('_')[1]
planta_desde = plantas[0]
planta_hasta = plantas[1]
planta_altura = plantas[2]
# lectura de datos
puntos = pd.read_csv(archivo_ruta,index_col='punto')
# busca gateway
n_Gtw = ''
for unpunto in puntos.index:
if unpunto.startswith('Gw'):
n_Gtw = unpunto
# perfil
xi = np.array(puntos['dist_'+n_Gtw])
yi = np.array(puntos['altitud'])
etiqueta = puntos.index
# plantaciones
conplantas = (xi > planta_desde) & (xi < planta_hasta)
plantacion = yi[conplantas]+planta_altura
# GRAFICA
# plantacion
plt.fill_between(xi[conplantas],
yi[conplantas],
plantacion,
color='lightgreen')
# perfil
xi = puntos['dist_'+n_Gtw]
yi = puntos['altitud']
plt.plot(xi,yi,color='brown')
# antenas
for unpunto in puntos.index:
xip = xi[unpunto]
yip = yi[unpunto]
if unpunto.startswith('Gw'):
xigtw = xip
yigtw = yip+alturaGw
plt.scatter(xip ,yigtw)
plt.annotate(unpunto,(xigtw,yigtw))
else:
xid = xip
yid = yip+alturapunto
plt.plot((xigtw,xid),(yigtw,yid),
color = 'green',
linestyle='dotted')
plt.scatter(xid ,yid)
plt.annotate(unpunto,(xid,yid), rotation=45)
plt.title('Linea :'+LineaObs)
plt.xlabel('distancia')
plt.ylabel('altura')
plt.grid()
# plt.axis('equal')
return(puntos)
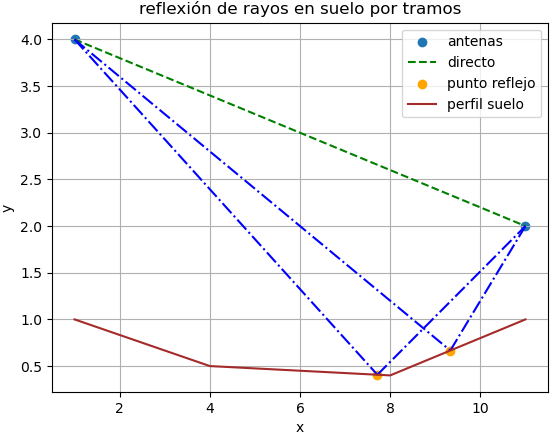
def revisa_fresnel(analiza_punto, archivo_ruta,alturaGw,alturapunto,
plantas,guarda=False,nFres =1,freq=915,muestras=41):
LineaObs = archivo_ruta.strip('.csv').split('_')[1]
planta_desde = plantas[0]
planta_hasta = plantas[1]
planta_altura = plantas[2]
# lambda Fresnel
lamb = 300/freq #300e6(m/s)/(freq*1e6)
puntos = pd.read_csv(archivo_ruta,index_col='punto')
# busca gateway
i_Gw = 0
i_punto = 0
n_Gtw = ''
i = 0
for unpunto in puntos.index:
if unpunto.startswith('Gw'):
n_Gtw = unpunto
if unpunto==analiza_punto:
i_punto = i
i = i+1
# perfil
xi = np.array(puntos['dist_'+n_Gtw])
yi = np.array(puntos['altitud'])
etiqueta = puntos.index
# plantaciones
conplantas = (xi > planta_desde) & (xi < planta_hasta)
plantacion = yi[conplantas]+planta_altura
# añade alturas de antenas
yi_ant = np.copy(yi) + alturapunto
yi_ant[i_Gw] = yi[i_Gw] + alturaGw
# Zona de Fresnel
if i_punto>0:
# linea directa
dy = yi_ant[i_punto]-yi_ant[i_Gw]
dx = xi[i_punto]-xi[i_Gw]
m = dy/dx
alpha = np.arctan(m)
yi_f = lambda x: m*x + yi_ant[i_Gw]
xi_D = np.linspace(xi[i_Gw],xi[i_punto],
muestras)
yi_D = yi_f(xi_D)
dist_D = np.sqrt(dx**2+dy**2)
# zona Fresnel 1
F1_up = np.zeros(len(xi_D))
F1_down = np.zeros(len(xi_D))
for i in range(0,len(xi_D),1):
d1 = xi_D[i]/np.cos(alpha)
d2 = dist_D-d1
F1 = np.sqrt(np.abs(nFres*lamb*d1*d2)/(d1+d2))
xiu = xi_D[i]-F1*np.sin(np.abs(alpha))
d1_u = xiu
d2_u = dist_D-d1_u
xid = xi_D[i]+F1*np.sin(np.abs(alpha))
d1_d = xid
d2_d = dist_D-d1_d
# Fresnel, formula
Fup = np.sqrt(np.abs(nFres*lamb*d1_u*d2_u)/(d1_u+d2_u))
Fdown = np.sqrt(np.abs(nFres*lamb*d1_d*d2_d)/(d1_d+d2_d))
# Linea directa +/- Fresnel
F1_up[i] = yi_D[i] + Fup*np.cos(alpha)
F1_down[i] = yi_D[i] - Fdown*np.cos(alpha)
else:
print('punto no encontrado',analiza_punto)
# SALIDA
# print('Radio Fresnel: ',np.max(F1u[i]))
# GRAFICA
plt.fill_between(xi[conplantas],yi[conplantas],
plantacion,color='lightgreen',
label = 'plantas')
plt.plot(xi,yi,label='perfil', color = "brown")
# antenas
plt.scatter(xi,yi_ant,label=
'Dispositivos',color='blue')
if i_punto>0:
# Fresnel
plt.plot(xi_D,yi_D,
label='Directo',color='orange',
linestyle = 'dashed')
plt.plot(xi_D,F1_down,
label='Fresnel1',color='orange')
plt.plot(xi_D,F1_up,
color='orange')
for i in range(0,len(xi),1):
plt.annotate(etiqueta[i],
(xi[i],yi_ant[i]),rotation=45)
plt.xlabel('distancia')
plt.ylabel('altura')
plt.title('Línea :'+LineaObs+', Enlace: '+n_Gtw+'-'+etiqueta[i_punto])
plt.legend()
plt.grid()
# plt.axis('equal')
# plt.show()
return(puntos)