Referencia: Chapra 17.1 p 466. Burden 8.1 p498, Mínimos cuadrados en Métodos numéricos
Las descripciones estadísticas de Rssi obtenidas en cada punto sobre una línea de propagación se usan para estimar el modelo de propagación para esa ruta.
La ecuación empírica con la que se realiza la primera estimación se modela como:
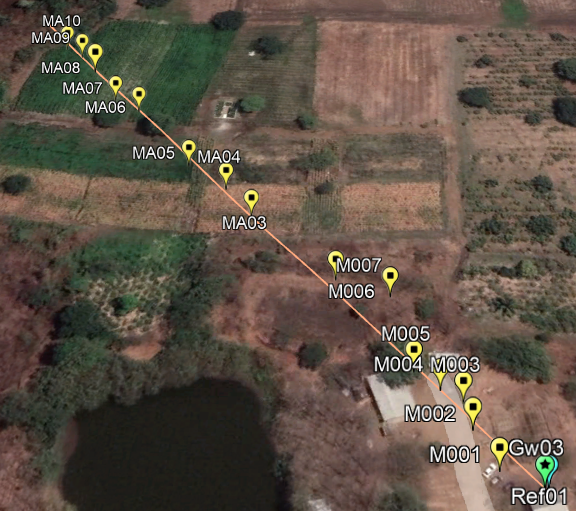
RSSI(d) = -10 \alpha \log_{10} (d) + P_{0} 1<d<LLa ruta de ejemplo sigue la dirección desde el gateway (Gw03) hacia una parcela de plantación de maiz (MA##) en la parte superior izquierda de la imagen. se añade la primera sección (M0##) para cubrir todo el segmento de propagación desde el gateway.
La imagen presentada representa la ruta c_MA, la letra "c" identifica el modelo de dispositivo usado (c: cápsula, m: módulo)
Los valores de Rssi_media en cada punto, tanto los enlaces de subida (up) y bajada (down), se linealizan por el método de los mínimos cuadrados usando la distancia como variable independiente.
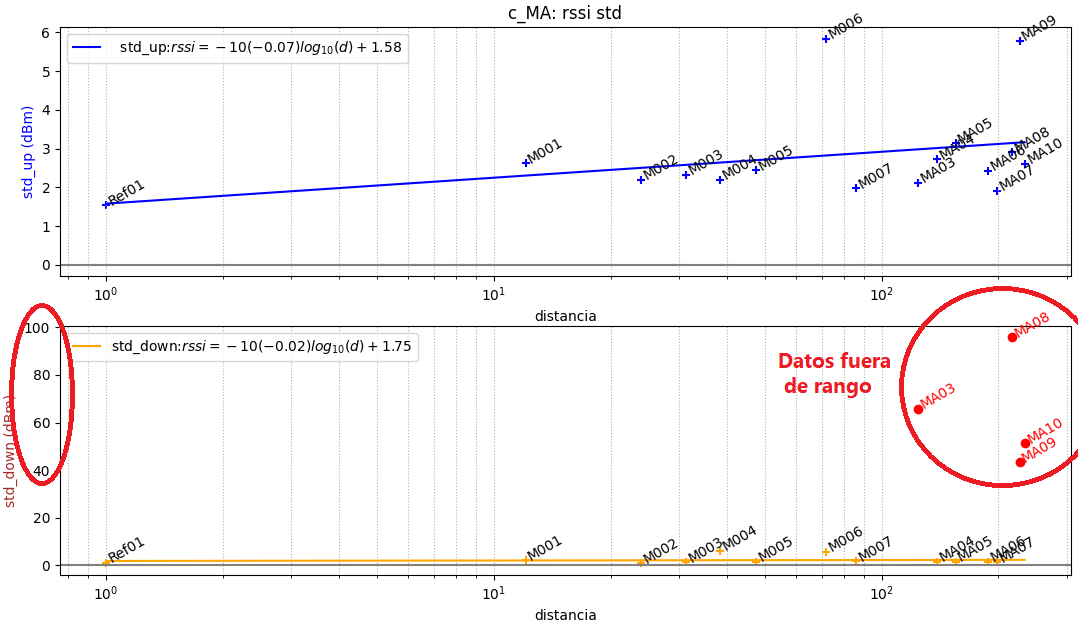
En cada punto Rssi se añaden las marcas '+' que representan media+std y media-std.
Mejorando el modelo
Las perdidas de señal desde el gateway aumentan con la distancia, siendo más grandes al final del segmento para el canal de bajada (down), mostrando una mayor desviación estándar (std) que afectan al modelo. Para discriminar los puntos que no representan una buena cobertura, se utiliza una gráfica de desviaciones estándar, donde los valores fuera de rango no se usan en el modelo.
La selección de los puntos que incluyen en el modelo se realiza en el archivo "rssi_c_MA_lista.txt"
punto,up,down M001,1,1 M002,1,1 M003,1,1 M004,1,1 M005,1,1 M006,1,1 M007,1,1 MA03,1,0 MA04,1,1 MA05,1,1 MA06,1,1 MA07,1,1 MA08,1,0 MA09,1,0 MA10,1,0 Ref01,1,1
En el archivo se muestra que los puntos MA03, MA08, MA09, MA10 no se usan para el enlace de bajada (down).
Se guarda el archivo de selección con la configuración, y se vuelve a ejecutar el algoritmo, y se obtiene el modelo mejorado de pérdidas de propagación:
ecuaciones:
ecuacion_up ecuacion_down
alpha 3.01 2.58
beta -21.37 -25.98
coef_correlacion -0.94 -0.93
coef_determinacion 0.88 0.86
eq_latex $ rssi = -10(3.01)log_{10}(d)-21.37 $ $ rssi = -10(2.58)log_{10}(d)-25.98 $
intervalox [1.0, 233.86] [1.0, 198.31]
error_medio 5.82 5.64
error_std 6.63 6.46
eqg_latex $ d = 10^{(-21.37 - rssi)/(10(3.01))} $ $ d = 10^{(-25.98 - rssi)/(10(2.58))} $
intervaloy [-97.87, -25.75] [-87.68, -26.99]
errorx_medio 43.32 37.68
errorx_std 56.01 47.11
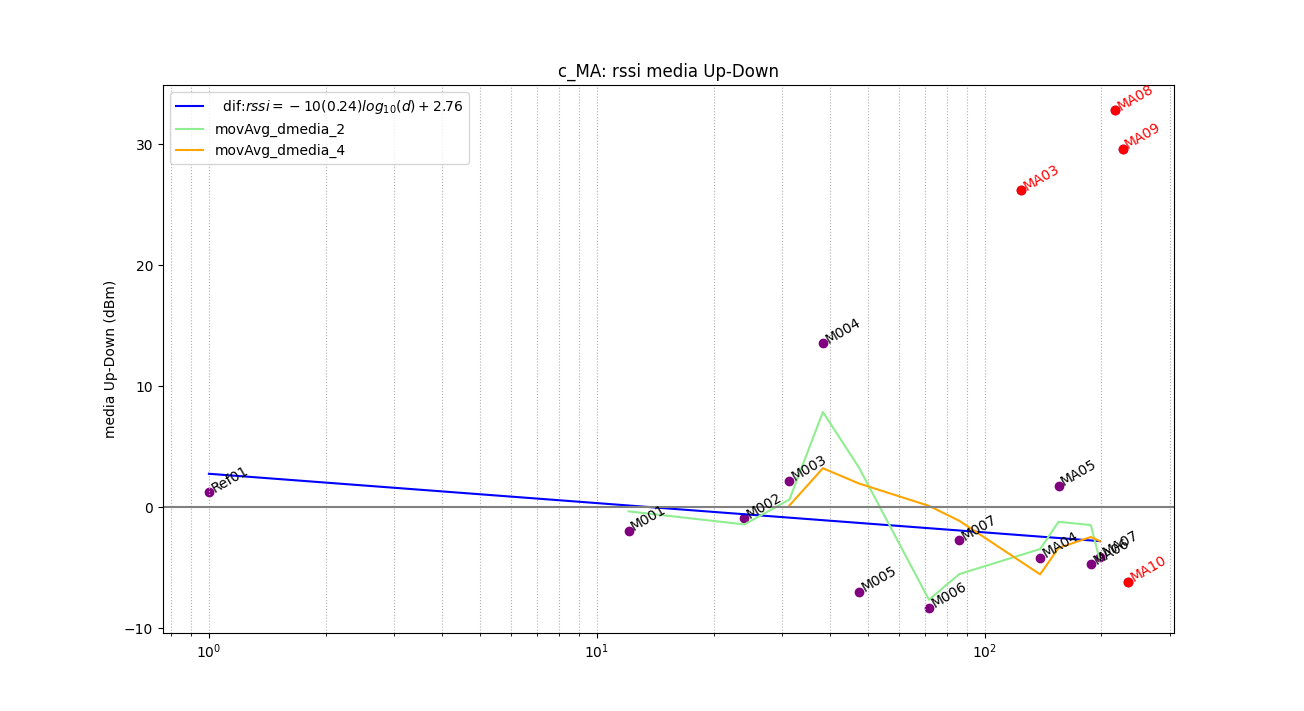
Una forma complementaria para observar los puntos "aberrantes" o fuera de rango es observar en cada punto la diferencia entre las medias del enlace de subida y el de bajada, donde se destaca que los valores se encuentran muy separados.
Las gráficas se realizan en bloques como funciones (def), a fin de activarlas o no durante el análisis detallado, además de incorporarlas a la librería girni_lorawan_lib.py
Las rutas posibles para este ejercicio se dan por medio de "carpeta_rsm", que es el directorio donde se encuentran los archivos con los detalles de cada punto.
Instrucciones en Python
Algunos parámetros se repiten entre algoritmos, por lo que se los ha separado en un archivo de texto 'variables_generales.txt'.
El contenido de las 'variables_generales.txt' se muestra a continuación:
{"carpeta_DB" : "BaseDatosJSON",
"medida" : "rssi",
"medida_unidad" : "dBm",
"medida_normal" : [-250,-1],
"medida_grafica" : [-100,-60],
"movAvg_cual" : [8,16],
"movAvg_color" : ["lightgreen","orange"],
"precision" : 2,
"gatewayDB" : {"uCfr//5zvhk=":"Gw01",
"uCfr//4dylc=":"Gw03"},
"arch_coord" : "coord_puntos.csv",
"carp_coord" : "coordenadas",
"zonaNum" : 17,
"zonaLetra" : "M",
"alturaGw" : 8,
"alturapunto": 1,
"nFres" : 1,
"freq" : 915,
"muestras" : 41,
"guarda" : 1
}
Con lo que las instrucciones en Python quedan como:
# Obtiene ecuación por mínimos cuadrados de una ruta # usando las medidas y distancia al gateway de cada punto # http://blog.espol.edu.ec/girni/ import numpy as np import pandas as pd import os import json import matplotlib.pyplot as plt import girni_lorawan_lib as girni # INGRESO carpeta_rsm = "resultado_c_Maiz_todo" arch_coord = "coord_puntos.csv" arch_var_general = "variables_generales.txt" # PROCEDIMIENTO # carga variables desde archivos with open(arch_var_general) as linea: texto = linea.read() var_gen = json.loads(texto) globals().update(var_gen) var_gen['movAvg_cual'] = [2,4] #cada cuantas muestras partes = carpeta_rsm.strip('/').split('_') arch_nombre = partes[1]+'_'+partes[2] def coordenadas_leer(arch_coord,carp_coord): ''' lista las coordenadas desde un archivo ubicado en la carpeta ''' if len(carp_coord)>0: carp_coord = carp_coord+'/' archivo_ruta = carp_coord+arch_coord arch_coord_existe = os.path.exists(archivo_ruta) if arch_coord_existe: puntos = pd.read_csv(archivo_ruta,index_col='punto') # busca gateway y ubicación de punto observado Gw_nombre = '' for unpunto in puntos.index: if unpunto.startswith('Gw'): Gw_nombre = unpunto dist_Gtw = puntos['dist_'+Gw_nombre] else: print(' ERROR: NO se encuentra el archivo...') print(' revise el ruta/nombre de archivo. ') return(dist_Gtw) dist_Gtw = coordenadas_leer(arch_coord,carp_coord) # crea lista archivos a usar si no existe arch_lista = medida+"_"+arch_nombre+"_lista.txt" archivo_ruta = carpeta_rsm + '/' + arch_lista archivoexiste = os.path.exists(archivo_ruta) if not(archivoexiste): puntoUsar_Colum = ['punto', 'up', 'down'] puntoUsar_Tipos = {'punto' : 'object', 'up' : 'int64', 'down' : 'int64'} puntoUsar = pd.DataFrame(columns = puntoUsar_Colum) puntoUsar = puntoUsar.astype(dtype = puntoUsar_Tipos) for unarchivo in os.listdir(carpeta_rsm): verifica = unarchivo.startswith('describe_') verifica = verifica and unarchivo.endswith('.csv') if verifica: partes = unarchivo.strip('.csv').split('_') unpunto = partes[2] puntodato = {'punto' : unpunto, 'up' : 1, 'down' : 1} puntodato = pd.DataFrame([puntodato]) puntoUsar = pd.concat([puntoUsar, puntodato], ignore_index = True) puntoUsar = puntoUsar.set_index('punto') puntoUsar.to_csv(archivo_ruta) if (archivoexiste): puntoUsar = pd.read_csv(archivo_ruta) puntoUsar = puntoUsar.set_index('punto') # Analiza cada punto en lista punto_columnas = ['codigopunto','dist_Gtw', medida+'_up',medida+'_up'+'_std', 'usar_up',medida+'_down', medida+'_down'+'_std', 'usar_down','dispositivo'] punto_tipos = {'codigopunto': 'object', 'dist_Gtw' : 'float64', medida+'_up' : 'float64', medida+'_up'+'_std': 'float64', 'usar_up' : 'int64', medida+'_down': 'float64', medida+'_down'+'_std' :'float64', 'usar_down' : 'int64', 'dispositivo': 'object'} punto_graf = pd.DataFrame(columns=punto_columnas) punto_graf = punto_graf.astype(dtype=punto_tipos) puntoSinDist =[] for unarchivo in os.listdir(carpeta_rsm): if unarchivo.startswith('describe_'): codigopunto = unarchivo.strip('.csv').split('_')[2] # lectura del archivo unresumen = carpeta_rsm+'/'+unarchivo descriptor = pd.read_csv(unresumen,index_col='Unnamed: 0') # Para Grafica condicion = (codigopunto in dist_Gtw.index) if condicion: undato = {'codigopunto': codigopunto, 'dist_Gtw' : dist_Gtw[codigopunto], medida+'_up' : descriptor['rssi_up']['mean'], medida+'_up'+'_std': descriptor['rssi_up']['std'], 'usar_up' : puntoUsar['up'][codigopunto], medida+'_down': descriptor['rssi_down']['mean'], medida+'_down'+'_std' :descriptor['rssi_down']['std'], 'usar_down' : puntoUsar['down'][codigopunto], 'dispositivo': descriptor['dispositivo'][0] } undato = pd.DataFrame([undato]) punto_graf = pd.concat([punto_graf,undato], ignore_index=True) else: puntoSinDist.append(codigopunto) punto_graf = punto_graf.set_index('codigopunto') punto_graf = punto_graf.sort_values('dist_Gtw' ) # selecciona datos dist = punto_graf['dist_Gtw'] usar_up = punto_graf['usar_up'] usar_down = punto_graf['usar_down'] # Eje x en log10() xi = np.array(dist) # enlace_up media_up = punto_graf[medida+'_up'] std_up = punto_graf[medida+'_up'+'_std'] media_up_techo = media_up + std_up media_up_piso = media_up - std_up # ecuacion Enlace up yi_up = np.array(media_up) eq_up = girni.linealiza_lstsq(xi[usar_up==1], yi_up[usar_up==1], digitos = precision) alpha_up = eq_up['alpha'] beta_up = eq_up['beta'] fd_up = lambda d: -10*alpha_up*(np.log10(d))+beta_up # Errores up respecto a ecuacion fi_up = fd_up(xi) dyi0std = eq_up['error_std'] punto_graf['fi_up'] = fi_up # enlace_down media_down = punto_graf[medida+'_down'] std_down = punto_graf[medida+'_down'+'_std'] media_down_techo = media_down + std_down media_down_piso = media_down - std_down # ecuación Enlace down yi_down = np.array(media_down) eq_down = girni.linealiza_lstsq(xi[usar_down==1], yi_down[usar_down==1], digitos = precision) alpha_down = eq_down['alpha'] beta_down = eq_down['beta'] fd_down = lambda d: -10*alpha_down*(np.log10(d))+beta_down # Errores down respecto a ecuacion fi_down = fd_down(xi) dyi1std = eq_down['error_std'] punto_graf['fi_down'] = fi_down resultado = {'tabla' : punto_graf, 'ecuacion_up' : eq_up, 'ecuacion_down': eq_down} ecuacion = resultado.copy() ecuacion.pop('tabla') ecuacion = pd.DataFrame(ecuacion) # SALIDA pd.options.display.float_format = '{:,.2f}'.format print(resultado['tabla']) print('\n ecuaciones: ') print(ecuacion) if len(puntoSinDist)>0: print('\n Error: Puntos que no registran distancia en coordenadas:') print(puntoSinDist) # graba la tabla resumen rssi unresumen = carpeta_rsm+'/'+medida+'_'+arch_nombre+'.csv' resultado['tabla'].to_csv(unresumen) def graf_gradiente(arch_nombre,punto_graf,ecuacion,var_gen): ''' grafica de un gradiente ''' escalabase = 10 # 10 escala = 'log' medida = var_gen['medida'] precision = var_gen['precision'] # enlace_up media_up = punto_graf[medida+'_up'] std_up = punto_graf[medida+'_up'+'_std'] media_up_techo = media_up + std_up media_up_piso = media_up - std_up yi_up = np.array(media_up) fi_up = punto_graf['fi_up'] # enlace_down media_down = punto_graf[medida+'_down'] std_down = punto_graf[medida+'_down'+'_std'] media_down_techo = media_down + std_down media_down_piso = media_down - std_down yi_down = np.array(media_down) fi_down = punto_graf['fi_down'] dist = punto_graf['dist_Gtw'] usar_up = punto_graf['usar_up'] usar_down = punto_graf['usar_down'] eq_up = ecuacion['ecuacion_up'] eq_down = ecuacion['ecuacion_down'] dyi0std = eq_up['error_std'] dyi1std = eq_down['error_std'] fig_gradnt,(graf_up,graf_down) = plt.subplots(2,1) if escala == 'log': graf_up.set_xscale(escala,base=escalabase) graf_down.set_xscale(escala,base=escalabase) # medida up +/- std graf_up.scatter(dist,media_up,color='blue',marker='o') graf_up.scatter(dist,media_up_techo,color='blue',marker='+') graf_up.scatter(dist,media_up_piso,color='blue',marker='+') # medida down +/- std graf_down.scatter(dist,media_down,color='orange',marker='o') graf_down.scatter(dist,media_down_techo,color='orange',marker='+') graf_down.scatter(dist,media_down_piso,color='orange',marker='+') # linealizado up graf_up.plot(dist,fi_up,label = ' up:'+eq_up['eq_latex'], color='blue') graf_up.plot(dist,fi_up+dyi0std, color='blue',linestyle='dotted') graf_up.plot(dist,fi_up-dyi0std, color='blue',linestyle='dotted') # linealizado down graf_down.plot(dist,fi_down,label = 'down:'+eq_down['eq_latex'], color='orange') graf_down.plot(dist,fi_down+dyi1std, color='orange',linestyle='dotted') graf_down.plot(dist,fi_down-dyi1std, color='orange',linestyle='dotted') # ajuste de eje y y_min = np.min([np.min(media_up_piso),np.min(media_down_piso)]) y_max = np.max([np.max(fi_up+dyi0std),np.max(fi_down+dyi1std)]) if y_min<-135: y_min = np.min([np.min([media_up]),np.min([media_down])]) graf_up.set_ylim(y_min,y_max) graf_down.set_ylim(y_min,y_max) # etiquetas up for unpunto in punto_graf.index: media_etiq = np.round(punto_graf[medida+'_up'][unpunto],precision) usarpunto = punto_graf['usar_up'][unpunto] etiqueta = unpunto #+str(media_etiq) xi = punto_graf['dist_Gtw'][unpunto] yi = punto_graf['rssi_up'][unpunto] if usarpunto: graf_up.annotate(etiqueta,(xi,yi), rotation=45) if not(usarpunto): graf_up.annotate(etiqueta,(xi,yi), rotation=45,color='red') graf_up.scatter(dist[unpunto],media_up[unpunto],color='red',marker='o') graf_up.scatter(dist[unpunto],media_up_techo[unpunto], color='red',marker='+') # etiquetas down for unpunto in punto_graf.index: media_etiq = np.round(punto_graf[medida+'_down'][unpunto],precision) usarpunto = punto_graf['usar_down'][unpunto] etiqueta = unpunto #+str(media_etiq) xi = punto_graf['dist_Gtw'][unpunto] yi = punto_graf['rssi_down'][unpunto] if usarpunto: graf_down.annotate(etiqueta,(xi,yi), rotation=45) if not(usarpunto): graf_down.annotate(etiqueta,(xi,yi), rotation=45,color='red') graf_down.scatter(dist[unpunto],media_down[unpunto],color='red',marker='o') graf_down.scatter(dist[unpunto],media_down_techo[unpunto], color='red',marker='+') graf_up.set_ylabel(medida+'_up (dBm)',color='blue') graf_up.set_title(arch_nombre+' '+medida) graf_up.legend() graf_up.grid(True,linestyle='dotted', axis='x', which='both') graf_down.set_xlabel('distancia') graf_down.set_ylabel(medida+'_down (dBm)',color='brown') graf_down.legend() graf_down.grid(True,linestyle='dotted', axis='x', which='both') return(fig_gradnt) def graf_diferencia_updown(arch_nombre,punto_graf,var_gen): '''Grafica de diferencias up-down ''' medida = var_gen['medida'] medida_unidad = var_gen['medida_unidad'] movAvg_cual = var_gen['movAvg_cual'] movAvg_color = var_gen['movAvg_color'] precision = var_gen['precision'] # analiza diferencias de medias y std -------------- punto_graf['usar_dif'] = punto_graf['usar_up']*punto_graf['usar_down'] usar_dif = punto_graf['usar_dif'] punto_graf['dif_mean'] = punto_graf['rssi_up']-punto_graf['rssi_down'] punto_graf['dif_std'] = punto_graf[medida+'_up_std']-punto_graf[medida+'_down_std'] punto_graf['dif_top'] = punto_graf['dif_mean']+punto_graf['dif_std'] punto_graf['dif_bottom'] = punto_graf['dif_mean']-punto_graf['dif_std'] dist = punto_graf['dist_Gtw'] usar_dif = punto_graf['usar_dif'] # linealiza diferencia xi_dif = np.array(punto_graf['dist_Gtw'][usar_dif==1]) yi_dif = np.array(punto_graf['dif_mean'][usar_dif==1]) eq_dif = girni.linealiza_lstsq(xi_dif,yi_dif,digitos = precision) alpha_dif = eq_dif['alpha'] beta_dif = eq_dif['beta'] fdistdif0 = lambda d: -10*alpha_dif*(np.log10(d))+beta_dif yi_dif0 = fdistdif0(xi_dif) ecua_dif = pd.DataFrame(eq_dif) # medias moviles en movAvg_cual[] serie_media = pd.Series(punto_graf['dif_mean'][usar_dif==1]) movAvg_dmedia = [] m = len(movAvg_cual) for j in range(0,m,1): k = movAvg_cual[j] movAvg_dmedia.append(serie_media.rolling(k).mean()) # Grafica diferencia escalabase = 10 # 10 escala = 'log' fig_dif_updown,graf_dmean = plt.subplots() if escala == 'log': graf_dmean.set_xscale(escala,base=escalabase) # diferencia medida up - down graf_dmean.scatter(dist,punto_graf['dif_mean'],color='purple',marker='o') graf_dmean.plot(xi_dif,yi_dif0,label = ' dif:'+eq_dif['eq_latex'],color='blue') graf_dmean.set_ylabel('media Up-Down (dBm)') # medida_up, medias móviles for j in range(0,m,1): k = str(movAvg_cual[j]) graf_dmean.plot(dist[usar_dif==1],movAvg_dmedia[j], label='movAvg_dmedia_'+k,color=movAvg_color[j]) # etiquetas i=0 for unpunto in punto_graf.index: media_etiq = np.round(punto_graf[medida+'_up'][unpunto],precision) etiqueta = unpunto # + ' ' +str(media_etiq) usar_dif = punto_graf['usar_dif'][unpunto] xi = punto_graf['dist_Gtw'][unpunto] yi = punto_graf.iloc[i]['dif_mean'] i = i+1 if usar_dif: graf_dmean.annotate(etiqueta,(xi,yi), rotation=30) if not(usar_dif): graf_dmean.annotate(etiqueta,(xi,yi),color='red',rotation=30) graf_dmean.scatter(xi,yi,color='red',marker='o') graf_dmean.axhline(0,color='grey') graf_dmean.grid(True,linestyle='dotted',axis='x', which='both') graf_dmean.legend() graf_dmean.set_title(arch_nombre+': '+medida+' media Up-Down') return(fig_dif_updown) def graf_gradiente_std(arch_nombre,punto_graf,medida,precision): ''' Grafica de std_up y std_down ------ ''' xi_std = np.array(punto_graf['dist_Gtw']) yi_std_up = np.array(punto_graf[medida+'_up_std']) yi_std_down = np.array(punto_graf[medida+'_down_std']) # linealiza std usar_up = punto_graf['usar_up'] usar_down = punto_graf['usar_down'] eq_std_up = girni.linealiza_lstsq(xi_std[usar_up==1], yi_std_up[usar_up==1], digitos = precision) alpha_std_up = eq_std_up['alpha'] beta_std_up = eq_std_up['beta'] fdiststd_up0 = lambda d: -10*alpha_std_up*(np.log10(d))+beta_std_up yi_std_up0 = fdiststd_up0(xi_std) eq_std_down = girni.linealiza_lstsq(xi_std[usar_down==1], yi_std_down[usar_down==1], digitos = precision) alpha_std_down = eq_std_down['alpha'] beta_std_down = eq_std_down['beta'] fdiststd_down0 = lambda d: -10*alpha_std_down*(np.log10(d))+beta_std_down yi_std_down0 = fdiststd_down0(xi_std) # grafica std escalabase = 10 # 10 escala = 'log' fig_grad_std,(graf_std_up,graf_std_down) = plt.subplots(2,1) if escala == 'log': graf_std_up.set_xscale(escala,base=escalabase) graf_std_down.set_xscale(escala,base=escalabase) graf_std_up.plot(xi_std,yi_std_up0, label = ' std_up:'+eq_std_up['eq_latex'], color='blue') # medida up +/- std graf_std_up.scatter(xi_std,yi_std_up,color='blue',marker='+') graf_std_down.scatter(xi_std,yi_std_down,color='orange',marker='+') # etiquetas i=0 for unpunto in punto_graf.index: media_etiq = np.round(punto_graf[medida+'_up'][unpunto],precision) etiqueta = unpunto # + ' ' +str(media_etiq) usar_up = punto_graf['usar_up'][unpunto] usar_down = punto_graf['usar_down'][unpunto] xi = punto_graf['dist_Gtw'][unpunto] yiu = punto_graf.iloc[i][medida+'_up_std'] yid = punto_graf.iloc[i][medida+'_down_std'] i = i+1 if usar_up: graf_std_up.annotate(etiqueta,(xi,yiu), rotation=30) if not(usar_up): graf_std_up.annotate(etiqueta,(xi,yiu),color='red',rotation=30) graf_std_up.scatter(xi,yiu,color='red',marker='o') if usar_down: graf_std_down.annotate(etiqueta,(xi,yid), rotation=30) if not(usar_down): graf_std_down.annotate(etiqueta,(xi,yid),color='red',rotation=30) graf_std_down.scatter(xi,yid,color='red',marker='o') graf_std_down.plot(xi_std,yi_std_down0, label = 'std_down:'+eq_std_down['eq_latex'], color='orange') # otras etiquetas graf_std_up.set_ylabel('std_up (dBm)', color='blue') graf_std_up.set_xlabel('distancia') graf_std_up.axhline(0,color='grey') graf_std_up.grid(True,linestyle='dotted', axis='x', which='both') graf_std_up.set_title(arch_nombre+': '+medida+' std') graf_std_up.legend() graf_std_down.set_ylabel('std_down (dBm)', color='brown') graf_std_down.set_xlabel('distancia') graf_std_down.axhline(0,color='grey') graf_std_down.grid(True,linestyle='dotted', axis='x', which='both') graf_std_down.legend() return(fig_grad_std) fig_gradnt = graf_gradiente(arch_nombre,punto_graf,ecuacion,var_gen) unarchivo = carpeta_rsm+'/ecuacion_'+arch_nombre+'.png' fig_gradnt.savefig(unarchivo) fig_dif_updown = graf_diferencia_updown(arch_nombre,punto_graf,var_gen) fig_grad_std = graf_gradiente_std(arch_nombre,punto_graf,medida,precision) plt.show() # pasa a programa principal