El efecto "sombra" añade el parámetro sector a la ecuación que se incorpora al resultado de la linealización en cada baliza.
La ecuación de una baliza se compone entonces de dos parámetros de selección: sector e intervalo. 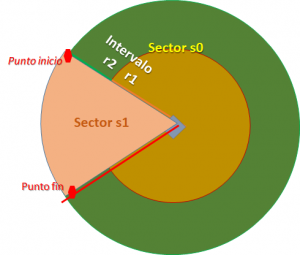
Como ilustración se muestra la figura que tiene tres partes o ecuaciones:
ecuacion['s0']['r1']
ecuacion['s0']['r2']
ecuacion['s1']['r0']
El primer parámetro para seleccionar la ecuación es el sector, 's0' y 's1', que en caso que sea un solo círculo se identifica como 's0'.
Dentro de cada sector, se mantiene el concepto de intervalos de distancia o radio. Se mantiene el concepto de la sección anterior, donde 'r0' corresponde a la linealización de todos los puntos en el sector. Cuando exiten sub-intervalos se usa 'r1', 'r2', etc para cada intervalo.
El número de ecuaciones corresponde al número de balizas y sectores establecidos para el análisis.
Se realizan cambios menores a la función pares_usar() de la librería girni para incorporar el parámetro sector, que al ser vacío '' funciona como fué descrito en las secciones anteriores.
pares_usar(tabla,baliza, analiza,unabaliza, unsector ='', medida = 'rssi', modo = 'rx')
También se actualizaron los nombres de los archivos de entrada y salida para diferenciar de los resultados anteriores y disponer de los archivos para comparar con los resultados del método que solo usa intervalos.
Algoritmo en Python
# LoRa-Multipunto, Rssi vs distancia con mínimos cuadrados # linealización Rssi vs log10(distancia) # por Sectores e intervalos , Graficas '2D' # Girni 2020-10-07 propuesta: edelros@espol.edu.ec import numpy as np import pandas as pd import json import matplotlib.pyplot as plt import girni_lora_libreria as girni # INGRESO # archivos de entrada modo = 'rx' medida = 'rssi' arch_medidaubica = 'rsmP06_'+medida+'Ubica01sector1.txt' # archivos de salida arch_ecuaciones = 'rsmP07_ecuacionSector01.json' arch_medUbAtrib = 'rsmP07_'+medida+'UbicaUsarSector01.txt' # Analizar por segmentos analiza = {'gtwRECT':{'analizar' : 1, 'sector_ref' : ['FIEC112','FCNM110'], 's0':{'atipico_std' : 1, 'frontera' : [300], 'atipInterv_std': [2,2], 'p_amplia': 2, 'grp' : ['RECT','FIEC'], 'tip' : ['punto'], 'LOS' : [1] }, 's1':{'atipico_std' : 1, 'frontera' : [], 'atipInterv_std': [2], 'p_amplia': 2, 'grp' : ['FIEC','FCNM'], 'tip' : ['punto'], 'LOS' : [1,0] } }, 'gtwFIEC':{'analizar' : 1, 'sector_ref' : [], 's0':{'atipico_std' : 1, 'frontera' : [190], 'atipInterv_std': [1,1], 'p_amplia': 4, 'grp' : ['FIEC','FCNM'], 'tip' : ['punto'], 'LOS' : [0,1] } }, 'gtwFCNM':{'analizar' : 1, 'sector_ref' : [], 's0':{'atipico_std' : 1, 'frontera' : [235.0], 'atipInterv_std': [2,2], 'p_amplia': 4, 'grp' : ['FIEC','FCNM'], 'tip' : ['punto'], 'LOS' : [1,0] } } } baliza = {'d1':'gtwRECT', 'd2':'gtwFIEC', 'd3':'gtwFCNM'} # Parámetros de grafica tipograf = '2D' # '2D','' sin grafica escala = 'log' # 'normal','log' escalabase = 10 # 10 casicero = 1e-4 precision = 2 intersectar = 1 # 0:Falso, 1: Verdadero # PROCEDIMIENTO # leer datos tabla = pd.read_csv(arch_medidaubica, index_col='etiqueta') tabla = pd.DataFrame(tabla) baliza_key = list(baliza.keys()) baliza_val = list(baliza.values()) # Resultados de análisis ecuacion = {} eq_graf = {} # analiza datos hacia una baliza for unabaliza in analiza: donde = baliza_val.index(unabaliza) cualbaliza = baliza_key[donde] # Parámetros analizar = analiza[unabaliza]['analizar'] if analizar: # Crea ecuacion por baliza ecuacion[unabaliza] = {'sector_rad':[]} eq_graf[unabaliza] = {} # sectores sectores = [] sector_ref = analiza[unabaliza]['sector_ref'] tabla['sector_'+cualbaliza] = 0 # 'todos' predeterminado # coordenadas baliza para identificar ángulo b_este = tabla['c_este'][unabaliza] b_norte = tabla['c_norte'][unabaliza] # sectores por puntos de referencia if len(sector_ref)>0: for cadapunto in sector_ref: p_este = tabla['c_este'][cadapunto] p_norte = tabla['c_norte'][cadapunto] dx = p_este - b_este dy = p_norte - b_norte theta = np.arctan2(dy,dx) if theta<0 and dx<0: theta = theta + 2*np.pi sectores.append(theta) sectores = np.array(sectores) ordenar = np.argsort(sectores) sectores = list(sectores[ordenar]) nsectores = len(sectores) # clasifica puntos por sector particular for cadapunto in tabla.index: dentrosector = 0 p_este = tabla['c_este'][cadapunto] p_norte = tabla['c_norte'][cadapunto] dx = p_este-b_este dy = p_norte-b_norte theta = np.arctan2(dy,dx) if theta<0 and dx<0: theta = theta + 2*np.pi for j in range(0,nsectores-1,1): if theta>sectores[j] and theta<sectores[j+1]: dentrosector = j+1 tabla.loc[cadapunto,'sector_'+cualbaliza] = dentrosector # ecuacion por baliza y sector ecuacion[unabaliza]['sector_rad'] = sectores nsectores = len(sectores) if nsectores == 0: nsectores = 1 for cadasector in range(0,nsectores,1): unsector = 's'+str(cadasector) ecuacion[unabaliza][unsector] = {} eq_graf[unabaliza][unsector] = {} # ecuación con todos los puntos como referencia [pares,par_etiqueta] = girni.pares_usar(tabla,baliza,analiza, unabaliza,unsector, medida,modo) xi = pares[:,0] yi = pares[:,1] n_xi = len(xi) # coeficiente de correlación correlacion = np.corrcoef(xi,yi)[0,1] # minimos cuadrados ecuacion0 = girni.linealiza_lstsq(xi,yi) # selecciona atipicos de todos los puntos atipico_std = analiza[unabaliza][unsector]['atipico_std'] alpha = ecuacion0['alpha'] beta = ecuacion0['beta'] fdist0 = lambda d: -10*alpha*(np.log10(d))+beta yi0 = fdist0(xi) dyi0std = ecuacion0['error_std'] dyi0 = yi - yi0 atipicos = np.abs(dyi0) >= dyi0std*atipico_std xi0_e = xi[atipicos] yi0_e = yi[atipicos] etiq0_e = par_etiqueta[atipicos] unintervalo = 'r0' # todos # para exportar hacia archivo o gráfica ecuacion[unabaliza][unsector] = {unintervalo: ecuacion0 } ecuacion[unabaliza][unsector][unintervalo]['correlacion'] = correlacion eq_graf[unabaliza][unsector] = {unintervalo: {'xi_graf' : xi, 'yi_graf' : yi, 'etiqueta' : par_etiqueta, 'linea' : yi0, 'atipicos' : [xi0_e,yi0_e], 'atip_etiq': etiq0_e} } # Intervalos radiales en sector intervalo = [np.min(xi),np.max(xi)] frontera = analiza[unabaliza][unsector]['frontera'] if len(frontera)>0: # revisar si frontera esta dentro intervalo frontera = np.array(frontera, dtype=float) revisar = (frontera>=np.min(xi)) & (frontera<=np.max(xi)) enintervalo = list(frontera[revisar]) intervalo.extend(enintervalo) intervalo = np.array(intervalo) ordenar = np.argsort(intervalo) intervalo = intervalo[ordenar] n_intervalo = len(intervalo) # analizar cada subintervalo p_inicio = 0 p_desde = 0 p_amplia = analiza[unabaliza][unsector]['p_amplia'] atipIntv_std = analiza[unabaliza][unsector]['atipInterv_std'] for i in range(0,n_intervalo-1,1): i_eq = 'r' + str(i+1) # puntos en subintervalo [a,b] a = intervalo[i] b = intervalo[i+1] subintervalo = (xi >= a) & (xi <= b) xi_sub = xi[subintervalo] yi_sub = yi[subintervalo] n_sub = len(xi_sub) etiq_sub = par_etiqueta[p_inicio:p_inicio + n_sub] # amplia sub-intervalo, mejora intersecta rectas detras = p_inicio retrocede = detras if detras > p_amplia: retrocede = p_amplia delante = n_xi - (p_inicio+n_sub)# -1) avanza = delante if delante >= p_amplia: avanza = p_amplia p_desde = p_inicio - retrocede p_hasta = p_inicio + (n_sub) + avanza p_inicio = p_inicio + (n_sub-1) # subintervalo, amplia puntos xi_a = xi[p_desde:p_hasta] yi_a = yi[p_desde:p_hasta] etiq_a = par_etiqueta[p_desde:p_hasta] # coeficiente de correlación correlacion1 = np.corrcoef(xi_a,yi_a)[0,1] # analiza subintervalo ecuacion1 = girni.linealiza_lstsq(xi_a,yi_a) ecuacion[unabaliza][unsector][i_eq] = ecuacion1 ecuacion[unabaliza][unsector][i_eq]['correlacion'] = correlacion1 # atipicos del subintervalo extendido alpha = ecuacion1['alpha'] beta = ecuacion1['beta'] fdist1 = lambda d: -10*alpha*(np.log10(d))+beta yi1 = fdist1(xi_a) dyi1std = ecuacion1['error_std'] atipicos = np.zeros(len(xi_a),dtype=bool) atipico_std = analiza[unabaliza][unsector]['atipInterv_std'][i] dyi1 = yi_a - yi1 if np.abs(dyi1std) > casicero: atipicos = np.abs(dyi1) >= dyi1std*atipico_std xi1_e = xi_a[atipicos] yi1_e = yi_a[atipicos] etiq1_e = etiq_a[atipicos] # para gráfica, atipicos sin extender puntos atipicos_sub = (xi1_e >= a) & (xi1_e<=b) xi_sub1_e = xi1_e[atipicos_sub] yi_sub1_e = yi1_e[atipicos_sub] etiq_sub1e = etiq1_e[atipicos_sub] eq_graf[unabaliza][unsector][i_eq] = {'atipicos': [xi_sub1_e,yi_sub1_e], 'atip_etiq':etiq_sub1e} # subintervalo sin atipicos if len(xi1_e)>0: atipicoNo = np.abs(dyi1) <= dyi1std*atipico_std xi2 = xi_a[atipicoNo] yi2 = yi_a[atipicoNo] etiq2 = etiq_a[atipicoNo] # coeficiente de correlación correlacion2 = np.corrcoef(xi2,yi2)[0,1] ecuacion2 = girni.linealiza_lstsq(xi2,yi2) # actualiza ecuación sin atipicos intervaloy intervalox = ecuacion1['intervalox'] ecuacion2['intervalox'] = intervalox.copy() alpha = ecuacion2['alpha'] beta = ecuacion2['beta'] fdist = lambda d: -10*alpha*(np.log10(d))+beta intervaloy = fdist(intervalox) ordenar = np.argsort(intervaloy) intervaloy = list(intervaloy[ordenar]) ecuacion2['intervaloy'] = intervaloy ecuacion[unabaliza][unsector][i_eq] = ecuacion2 ecuacion[unabaliza][unsector][i_eq]['correlacion'] = correlacion2 # Revisar frontera entre subintervalos, # calcula intersección de rectas como nueva frontera interv_calc = np.copy(intervalo) if len(intervalo) >2 and intersectar==1 : for i in range(0,n_intervalo-2,1): ai = 'r' + str(i+1) bi = 'r' + str(i+2) ma = ecuacion[unabaliza][unsector][ai]['alpha'] ba = ecuacion[unabaliza][unsector][ai]['beta'] mb = ecuacion[unabaliza][unsector][bi]['alpha'] bb = ecuacion[unabaliza][unsector][bi]['beta'] # punto de intersección o cruce cruzanx = 10**((bb-ba)/(10*(mb-ma))) dfrontera = frontera - cruzanx # cruce dentro de intervalo de ecuacion if cruzanx > intervalo[-1]: cruzanx = intervalo[-1] if cruzanx < intervalo[0]: cruzanx = intervalo[0] interv_calc[i+1] = cruzanx # para grafica evalua cada subintervalo sin atipicos n_interv_calc = len(interv_calc) for i in range(0,n_interv_calc-1,1): i_eq = 'r'+str(i+1) a = interv_calc[i] b = interv_calc[i+1] subintervalo = (xi >= a) & (xi <= b) xi_sub = xi[subintervalo] yi_sub = yi[subintervalo] xi_graf = np.copy(xi[subintervalo]) if not(a in xi_graf): xi_graf = np.concatenate(([a],xi_graf),axis=0) if not(b in xi_graf): xi_graf = np.concatenate((xi_graf,[b]),axis=0) # Evalua subintervalo con la ecuacion sin atipicos alpha = ecuacion[unabaliza][unsector][i_eq]['alpha'] beta = ecuacion[unabaliza][unsector][i_eq]['beta'] fdist = lambda d: -10*alpha*(np.log10(d))+beta yi1_sub = fdist(xi_sub) yi_graf = fdist(xi_graf) eq_graf[unabaliza][unsector][i_eq]['xi_graf'] = xi_graf eq_graf[unabaliza][unsector][i_eq]['yi_graf'] = yi_graf a = np.round(np.min([xi_graf]),precision) b = np.round(np.max([xi_graf]),precision) ecuacion[unabaliza][unsector][i_eq]['intervalox'] = [a,b] ay = np.round(np.min([yi_graf]),precision) by = np.round(np.max([yi_graf]),precision) ecuacion[unabaliza][unsector][i_eq]['intervaloy'] = [ay,by] # SALIDA for unabaliza in ecuacion: for unsector in ecuacion[unabaliza]: if unsector == 'sector_rad': print('baliza: ',unabaliza) print(' sectores radianes: ',ecuacion[unabaliza]['sector_rad']) if unsector != 'sector_rad': for i_eq in ecuacion[unabaliza][unsector]: unintervalo = ecuacion[unabaliza][unsector][i_eq]['intervalox'] unintervaloy = ecuacion[unabaliza][unsector][i_eq]['intervaloy'] error_medio = ecuacion[unabaliza][unsector][i_eq]['error_medio'] error_std = ecuacion[unabaliza][unsector][i_eq]['error_std'] eq_latex = ecuacion[unabaliza][unsector][i_eq]['eq_latex'] errorx_medio = ecuacion[unabaliza][unsector][i_eq]['errorx_medio'] errorx_std = ecuacion[unabaliza][unsector][i_eq]['errorx_std'] correlacion = ecuacion[unabaliza][unsector][i_eq]['correlacion'] print(' [sector][intervalo]: ',unsector,',',i_eq) print(' ' + eq_latex) print(' ','intervalox: ',np.round(unintervalo,precision)) print(' ','intervaloy: ',np.round(unintervaloy,precision)) print(' correlación: ',np.round(correlacion,precision)) print(' |error_rssi| promedio: ',np.round(error_medio,precision), ' , std:',np.round(error_std,precision)) print(' |error_dist| promedio: ',np.round(errorx_medio,precision), ' , std:',np.round(errorx_std,precision)) print() # salida hacia archivo with open(arch_ecuaciones, 'w') as outfile: json.dump(ecuacion, outfile) tabla.to_csv(arch_medUbAtrib) # GRAFICA # Referencias para gráfica grupo = ['FIEC' ,'FCNM' ,'RECT','CIRC'] colores = ['green','orange','grey','magenta'] tipo = ['punto','1m' ,'gtw','dispositivo'] marcas = [ 'o','D' ,'D' ,'*' ] if tipograf=='2D': for unabaliza in ecuacion: for unsector in ecuacion[unabaliza]: if not(unsector=='sector_rad'): figura,grafica = plt.subplots() if escala == 'log': grafica.set_xscale(escala,base=escalabase) # todos los puntos unintervalo = 'r0' xi = eq_graf[unabaliza][unsector][unintervalo]['xi_graf'] yi = eq_graf[unabaliza][unsector][unintervalo]['yi_graf'] etiqueta = eq_graf[unabaliza][unsector][unintervalo]['etiqueta'] grafica.scatter(xi,yi,marker='.') m = len(xi) for i in range(0,m,1): grafica.annotate(etiqueta[i],(xi[i],yi[i])) # linealizado con todos los puntos fdtxt = ecuacion[unabaliza][unsector][unintervalo]['eq_latex'] yi0 = eq_graf[unabaliza][unsector][unintervalo]['linea'] a = np.round(np.min([xi]),precision) b = np.round(np.max([xi]),precision) eq_texto = fdtxt +' ; ['+ str(a) +','+ str(b)+']' grafica.plot(xi,yi0,label=eq_texto,linestyle='dotted') # linealizado por subintervalo eq_interv = list(ecuacion[unabaliza][unsector].keys()) eq_interv.pop(0) n_intervalo = len(eq_interv) for i_eq in eq_interv: fdtxt = ecuacion[unabaliza][unsector][i_eq]['eq_latex'] grtxt = ecuacion[unabaliza][unsector][i_eq]['eqg_latex'] xi_graf = eq_graf[unabaliza][unsector][i_eq]['xi_graf'] yi_graf = eq_graf[unabaliza][unsector][i_eq]['yi_graf'] a = np.round(np.min([xi_graf]),precision) b = np.round(np.max([xi_graf]),precision) eq_texto = fdtxt+' ; ['+str(a)+','+str(b)+']' grafica.plot(xi_graf,yi_graf, label=eq_texto) # atipicos marcados en subintervalo [xi1_e,yi1_e] = eq_graf[unabaliza][unsector][i_eq]['atipicos'] etiq1_e = eq_graf[unabaliza][unsector][i_eq]['atip_etiq'] grafica.scatter(xi1_e,yi1_e, color='red') m = len(etiq1_e) for i in range(0,m,1): grafica.annotate(etiq1_e[i], (xi1_e[i],yi1_e[i]), color='red') # lineas de frontera grafica.axvline(a, color='lightblue') valor_frontera = str(np.round(a,precision)) grafica.annotate(valor_frontera, (a,np.max([yi,yi0])), color='lightblue') grafica.axvline(b, color='lightblue') valor_frontera = str(np.round(b,precision)) grafica.annotate(valor_frontera, (b,np.max([yi,yi0])), color='lightblue') # etiquetas y títulos grafica.legend() grafica.set_ylabel(medida+'_'+modo) grafica.set_xlabel('distancia') grafica.grid(True,linestyle='dotted', axis='x', which='both') untitulo = unabaliza+'_'+unsector+': ' untitulo = untitulo+medida+'_'+modo+' vs distancia' grafica.set_title(untitulo) plt.show()
