Ejercicio: 3Eva_IT2019_T2 Integral con interpolación
El ejercicio considera dos partes: interpolación e integración
a. Interpolación
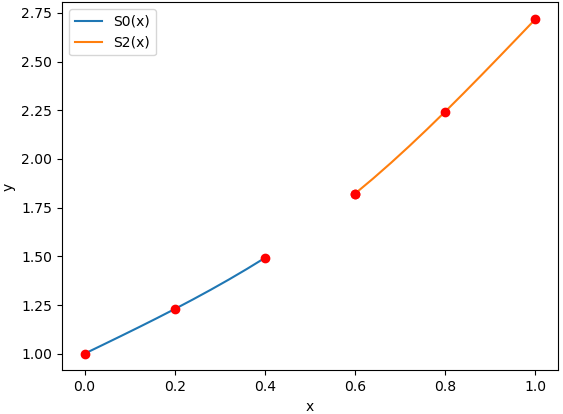
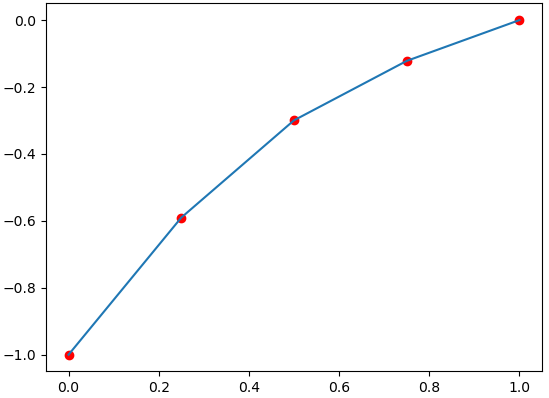
Se requiere aproximar la función usando tres puntos. Para comprender la razón del método solicitado, se compara la función con dos interpolaciones:
a.1 Lagrange
a.2 Trazador cúbico sujeto

Observando la gráfica se aclara que en éste caso, una mejor aproximación se obtiene con el método de trazador cúbico sujeto. Motivo por lo que el tema tiene un peso de 40/100 puntos
Los valores a considerar para la evaluación son:
puntos referencia xi,yi:
[0. 0.78539816 1.57079633]
[ 0. 0.62426595 -0.97536797]
derivadas en los extremos:
3.141592653589793
0.6929852019184021
Polinomio de Lagrange
-1.80262534301178*x**2 + 2.21061873102778*x
Trazadores cúbicos sujetos
[0. 0.78539816]
-0.548171611756137*x**3 - 2.55744517923506*x**2 + 3.14159265358979*x
[0.78539816 1.57079633]
4.66299098804068*x**3 - 14.8359577843727*x**2 + 12.7851139029174*x - 2.52466795930204
------------------
Valores calculados para Trazadores cúbicos sujetos:
Matriz A:
[[-0.26179939 -0.13089969 0. ]
[ 0.78539816 3.14159265 0.78539816]
[ 0. 0.13089969 0.26179939]]
Vector B:
[ 2.34675256 -16.9893436 2.72970237]
coeficientes S:
[-5.11489036 -7.69808822 14.27573913]
coeficientes a,b,c,d
[-0.54817161 4.66299099]
[-2.55744518 -3.84904411]
[ 3.14159265 -1.89005227]
[0. 0.62426595]
b. Integración
Como forma de comparacíon de resultados, se requiere integrar con varios métodos para comparar resultados y errores.
b.1 Integración con Cuadratura de Gauss, usando el resultado de trazador cúbico.
Se integra en cada tramo de cada polinomio:
Trazadores cúbicos sujetos
[0. 0.78539816]
-0.548171611756137*x**3 - 2.55744517923506*x**2 + 3.14159265358979*x
Se obtienen los puntos del método de cuadratura desplazados en el rango:
xa: 0.16597416116944688
xb: 0.6194240022280014
area: 0.5037962958529855
Para el segundo tramo:
[0.78539816 1.57079633]
4.66299098804068*x**3 - 14.8359577843727*x**2 + 12.7851139029174*x - 2.52466795930204
xa: 0.9513723245668951
xb: 1.4048221656254496
area: -0.2706563884589365
Con lo que el integral total es:
Integral total: 0.23313990739404894
b.2 Integración analítica
\int_0^{\pi /2}sin(\pi x) dx
u = πx
du/dx = π
dx = du/π
se convierte en:
\frac{1}{\pi}\int sin(u) du
\frac{1}{\pi}(-cos(u))
volviendo a la variable x:
\frac{1}{\pi}(-cos(\pi x)) \Big\rvert_{0}^{\frac{\pi}{2}}
-\frac{1}{\pi}(cos(\pi \frac{\pi}{2})-cos(\pi(0))) = 0.24809580527879377
c. Estimación del error
Se restan los resultados de las secciones b.1 y b.2
error = |0.24809580527879377 – 0.23313990739404894 |
error = 0.014955897884744829
Algoritmo en Python
separado por literales
# 3Eva I T 2019 Interpola e Integra
import numpy as np
import sympy as sym
import matplotlib.pyplot as plt
def interpola_lagrange(xi,yi):
'''
Interpolación con método de Lagrange
resultado: polinomio en forma simbólica
'''
# PROCEDIMIENTO
n = len(xi)
x = sym.Symbol('x')
# Polinomio
polinomio = 0
for i in range(0,n,1):
# Termino de Lagrange
termino = 1
for j in range(0,n,1):
if (j!=i):
termino = termino*(x-xi[j])/(xi[i]-xi[j])
polinomio = polinomio + termino*yi[i]
# Expande el polinomio
polinomio = polinomio.expand()
return(polinomio)
def traza3sujeto(xi,yi,u,v):
'''
Trazador cúbico sujeto, splines
resultado: polinomio en forma simbólica
'''
n = len(xi)
# Valores h
h = np.zeros(n-1, dtype=float)
# Sistema de ecuaciones
A = np.zeros(shape=(n,n), dtype=float)
B = np.zeros(n, dtype=float)
S = np.zeros(n-1, dtype=float)
# coeficientes
a = np.zeros(n-1, dtype=float)
b = np.zeros(n-1, dtype=float)
c = np.zeros(n-1, dtype=float)
d = np.zeros(n-1, dtype=float)
polinomios=[]
if (n>=3):
for i in range(0,n-1,1):
h[i]=xi[i+1]-xi[i]
A[0,0] = -h[0]/3
A[0,1] = -h[0]/6
B[0] = u-(yi[1]-yi[0])/h[0]
for i in range(1,n-1,1):
A[i,i-1] = h[i-1]
A[i,i] = 2*(h[i-1]+h[i])
A[i,i+1] = h[i]
B[i] = 6*((yi[i+1]-yi[i])/h[i] - (yi[i]-yi[i-1])/h[i-1])
A[n-1,n-2] = h[n-2]/6
A[n-1,n-1] = h[n-2]/3
B[n-1] = v-(yi[n-1]-yi[n-2])/h[n-2]
# Resolver sistema de ecuaciones
S = np.linalg.solve(A,B)
# Coeficientes
for i in range(0,n-1,1):
a[i]=(S[i+1]-S[i])/(6*h[i])
b[i]=S[i]/2
c[i]=(yi[i+1]-yi[i])/h[i]-(2*h[i]*S[i]+h[i]*S[i+1])/6
d[i]=yi[i]
# polinomio en forma simbólica
x=sym.Symbol('x')
polinomios=[]
for i in range(0,n-1,1):
ptramo = a[i]*(x-xi[i])**3 + b[i]*(x-xi[i])**2 + c[i]*(x-xi[i])+ d[i]
ptramo = ptramo.expand()
polinomios.append(ptramo)
parametros = [A,B,S,a,b,c,d]
return(polinomios, parametros)
# INGRESO
f = lambda x: np.sin(np.pi*x)
muestrasf = 20
a = 0
b = np.pi/2
# Derivadas en los extremos
u = np.pi*np.cos(np.pi*a)
v = np.pi*np.cos(np.pi*b)
muestras = 3
# literal a
# PROCEDIMIENTO
xif = np.linspace(a,b,muestrasf)
yif = f(xif)
xi = np.linspace(a,b,muestras)
yi = f(xi)
# Usando Lagrange
x = sym.Symbol('x')
pL = interpola_lagrange(xi,yi)
pxL = sym.lambdify(x,pL)
pxiL = pxL(xif)
# Trazador cúbico sujeto
pS, parametros = traza3sujeto(xi,yi,u,v)
pxiS = np.zeros(muestrasf,dtype=float)
# Evalua trazadores cúbicos sujetos
i=0
ap = xi[i]
bp = xi[i+1]
poli = sym.lambdify(x, pS[i])
for j in range(0,muestrasf,1):
punto = xif[j]
if (punto>bp):
i = i+1
ap = xi[i]
bp = xi[i+1]
poli = sym.lambdify(x,pS[i])
pxiS[j] = poli(punto)
# SALIDA
print('puntos referencia xi,yi: ')
print(xi)
print(yi)
print('derivadas en los extremos: ',u,v)
print('Polinomio de Lagrange')
print(pL)
print('Trazadores cúbicos sujetos')
n = len(xi)
for i in range(0,n-1,1):
print(xi[i:i+2])
print(pS[i])
# Parametros de Trazadores cúbicos sujetos
print('Matriz A: ')
print(parametros[0])
print('Vector B: ')
print(parametros[1])
print('coeficientes S: ')
print(parametros[2])
print('coeficienetes a,b,c,d')
print(parametros[3])
print(parametros[4])
print(parametros[5])
print(parametros[6])
# Gráficas
plt.plot(xif,yif, label='funcion')
plt.plot(xi,yi,'o', label='muestras')
plt.plot(xif,pxiL, label='p(x)_Lagrange')
plt.plot(xif,pxiS, label='p(x)_Traza3Sujeto')
plt.legend()
plt.xlabel('x')
plt.show()
# literal b
# cuadratura de Gauss de dos puntos
def integraCuadGauss2p(funcionx,a,b):
x0 = -1/np.sqrt(3)
x1 = -x0
xa = (b+a)/2 + (b-a)/2*(x0)
xb = (b+a)/2 + (b-a)/2*(x1)
area = ((b-a)/2)*(funcionx(xa) + funcionx(xb))
print('xa: ',xa)
print('xb: ',xb)
print('area: ', area)
return(area)
# INGRESO
f0 = sym.lambdify(x,pS[0])
f1 = sym.lambdify(x,pS[1])
# Procedimiento
I0 = integraCuadGauss2p(f0,xi[0],xi[1])
I1 = integraCuadGauss2p(f1,xi[1],xi[2])
It = I0+I1
# SALIDA
print('Integral 1: ', I0)
print('Integral 2: ', I1)
print('Integral total: ',It)